この講義の内容は下記のYouTubeサイト(生命科学を専攻する学生の為の分子生物学講義)で動画としても視聴できます
https://www.youtube.com/watch?v=LuS5L60wZdU&list=PL_B52Q_vHW1Y0YgqMBs_HZXBbtISm3L7B&index=12
1. この講義には下記のような内容が含まれています。25枚の図を含んでいます。
この講義では核ゲノムにあるイントロンの起源と進化について解説をしていきます。
1) 真核細胞におけるGroup I、Group IIおよびspliceosome 型イントロンの細胞内分布。
2) 真正細菌ゲノムのイントロンの種類と古細菌ゲノムのイントロンの種類。
3) イントロン内のORFの翻訳
4) Group IIとspliceosomeイントロンのスプライシング反応の類似性
5) Group IとGroup IIイントロンのスプライシング反応の比較
6) 小核RNAが形成する立体構造とGroup IIイントロンの立体構造の類似性
7) Group IIにコードされるORFタンパク質が保持する酵素活性
8) 初期の真核細胞におけるGroup IIイントロンに由来するspliceosomeイントロンの進化。
9) Group IIイントロンのRetrohoming機構と非LTRレトロトランスポゾンの転移機構との類似性。
10) Group IイントロンのHoming機構
11) Homing酵素の認識特異性
12) 細菌の接合によるゲノム転移。
Key Words: グループI、グループII、イントロン、スプライセオソーム、イントロンORF、真正細菌、古細菌、葉緑体、ミトコンドリア、リボザイム、snRNA、U1RNA、U6RNA、IBS、EBS、非LTRレトロトランスポゾン、逆転写、レトロホーミング、イントロン転移, Group I, Group II, intron, spliceosomal, intronic ORF, eubacteria, archaea, chloroplast, mitochondria, ribozyme, snRNA, U1RNA, U6RNA, IBS, EBS, non-LTR retrotransposon, reverse transcription, retrohoming, intron transfer
2. 真核細胞にあるイントロンの分布図

この図は真核生物の細胞を表しています。核のゲノムの中には spliceosomeと呼ばれるイントロンの切り出し装置によって 切り出されるようなイントロンが多数あります。ヒストンタンパクをコードする以外は,全てのタンパクコード遺伝子には,少なくとも一つのイントロンが含まれています。
また,ミトコンドリアのゲノムの中には Group Iイントロン、Group IIイントロンと呼ばれるような 2種類のイントロンがあります。葉緑体のゲノムの中にも Group Iイントロン、Group IIイントロンと呼ばれる 2種類のイントロンが組み込まれています。
一方,これらオルガネラのゲノムにコードされている遺伝子の中には,spliceosomeで切り出されるようなイントロンはありません。
3. 核ゲノムにコードされているタンパク遺伝子のsplicing
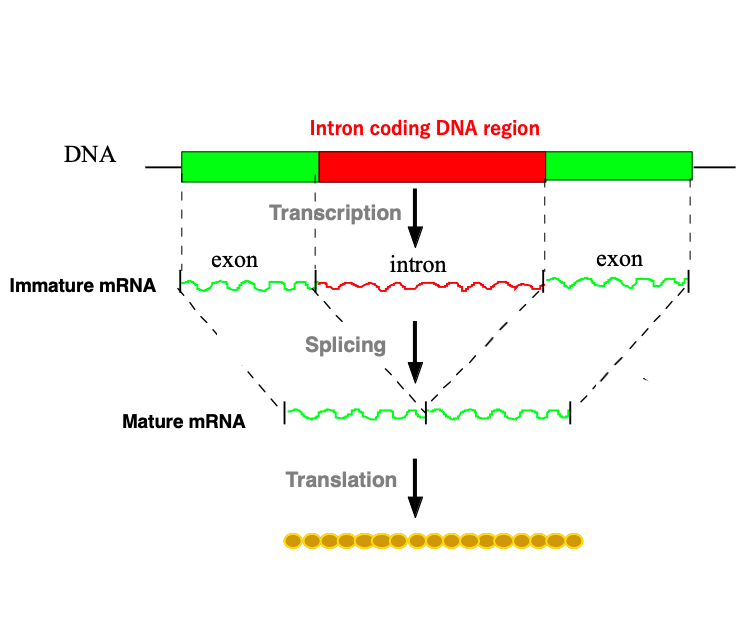
この図は,真核生物の核ゲノムにコードされているタンパク遺伝子のsplicingの概要を示しています。
核内で転写されたばかりのmRNAはイントロンを含みます。このような状態のmeRNAは,Immature messenger RNAと呼ばれます。このイントロンを取り除き,残ったexon領域を結合させる装置がSpliceosomeです。
Spliceosomeは, U1, U2, U3, U4, U5, U6と呼ばれる6種類の短いRNAと100種類以上のタンパクの複合体です。Splicing反応が終わったmessenger RNAはmature mRNAと呼ばれます。mature mRNAは核膜孔から細胞質に移送され,そこで翻訳されます。
4. 細菌におけるイントロンの分布
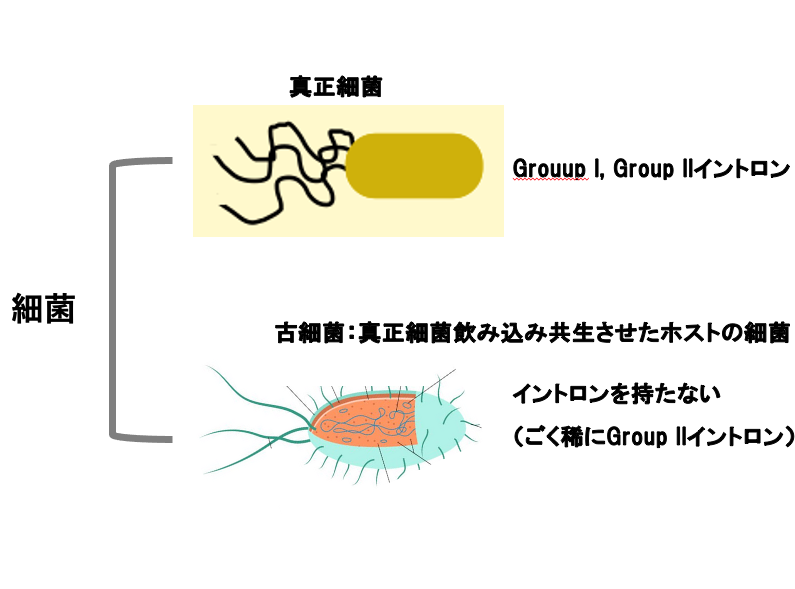
全ての細菌のゲノムの中には、spliceosomeで切り出されるタイプのイントロはありませんが、Group IやGroup IIイントロンが存在します。
細菌は大きく、真正細菌と古細菌に分類されます。古細菌は真正細菌を飲み込み,共生を経て真核生物が誕生するという進化上の一大イベントのホスト細胞としてよく知られています。
古細菌にはほとんどイントロがありません。ごく稀にGroup IIイントロンが発見されるだけです。一方、真正細菌にはGroup IやGroup IIイントロンが多数見つかっています。
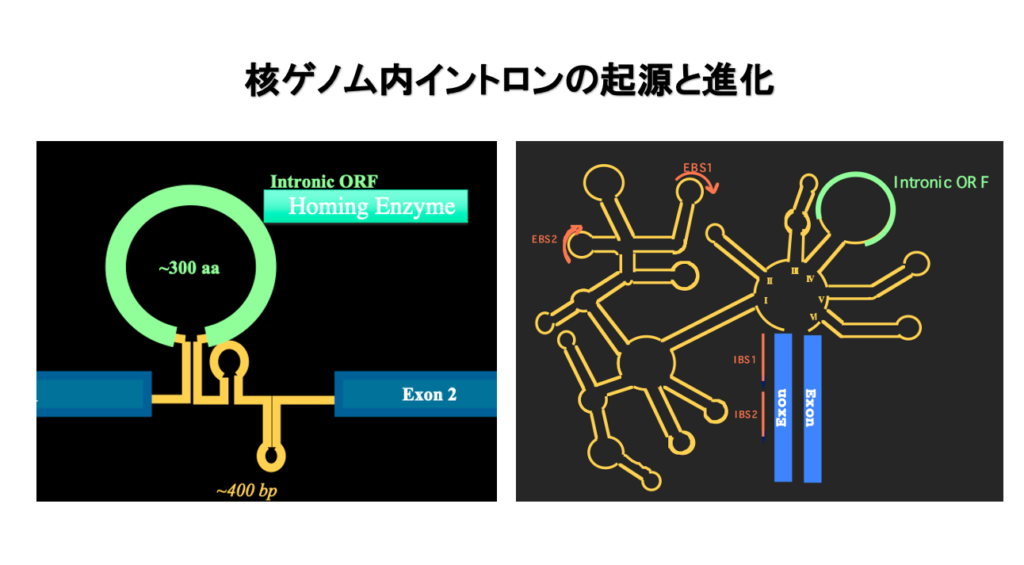
5. 立体構造と内図ORFを持つイントロン: Group Iイントロン, Group II
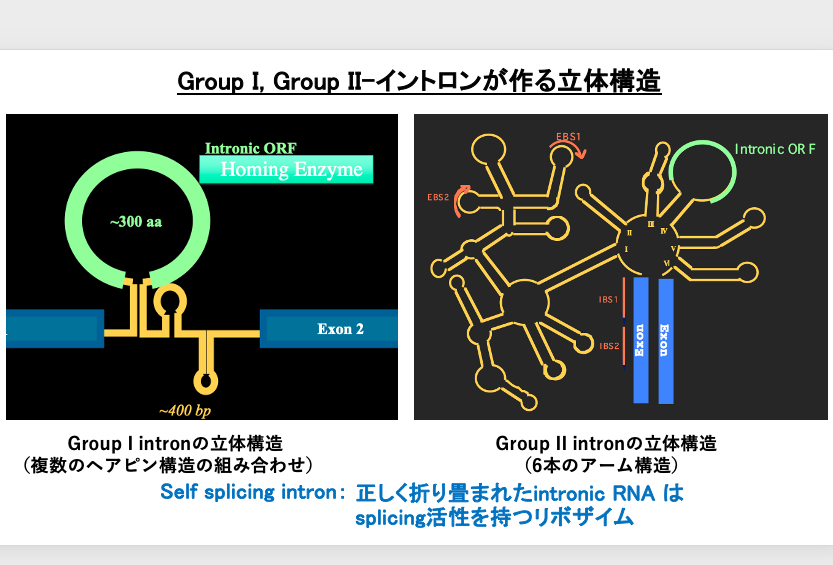
まずはGroup IイントロンとGroup IIイントロンが作る立体構造の特徴についてお話をしていきます。spliceosomeが切り出す真核生物のイントロンでは その切り出されるイントロン部分は,特別な立体構造を持っていません。
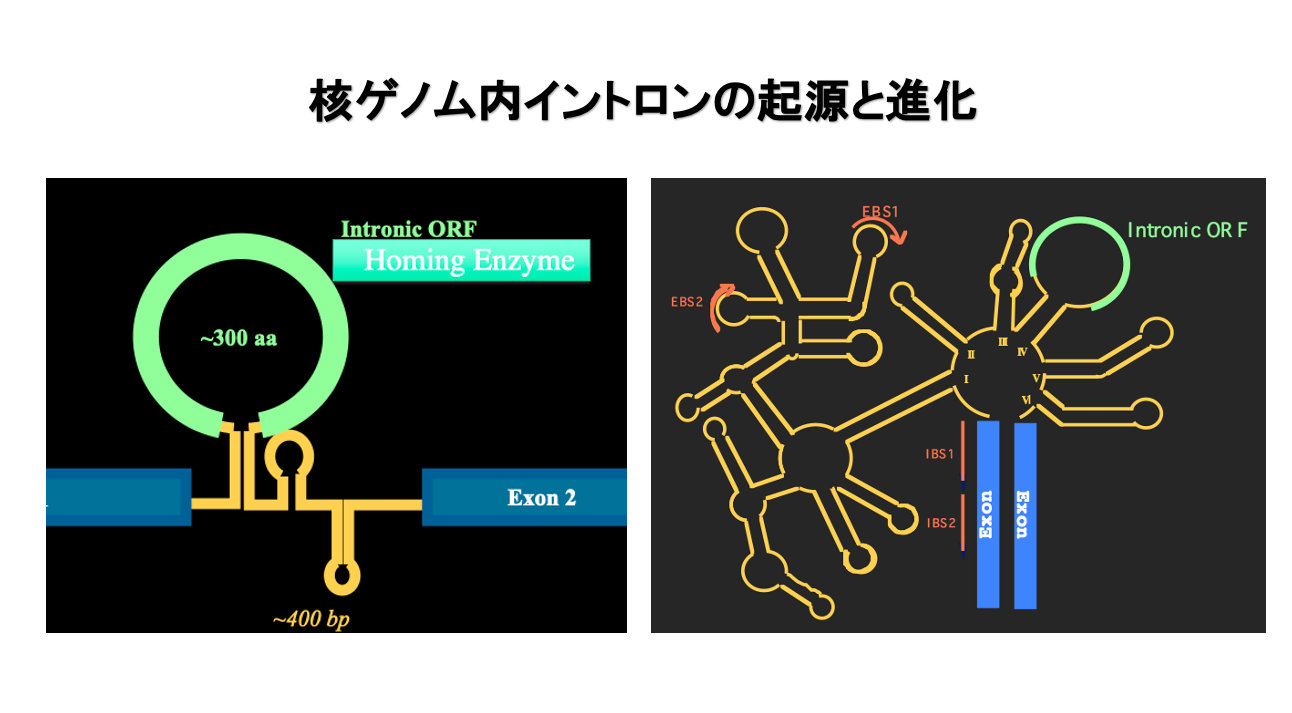
一方,Group IイントロンやGroup IIイントロンでは, 切り出されるイントロン部分が非常に特徴的な立体構造を作ります。 Group Iイントロンでは,このようなヘアピン構造がいくつか組み合わさった立体構造を持っています。ヘアピン構造の数は一定していません。また特徴的なのは,どこかのヘアピンのループ内部にタンパクをコードするよopen reading frame (ORF)が見出されることです。イントロンの中にあるので intronic ORFとこの図では書いてあります。
このintronic ORFにコードされているのは ホーミングエンザイムと呼ばれる活性を持つようなタンパクです。一方,Group IIイントロンは6つのアーム構造が組み合わさってできています。 1番目のアームは非常に大きいですね。2番目、3番目、4、5、6番目という風に 6つのアームが組み合わさって出来たのがGroup IIイントロンということになります。 また4番目のアームの中には intronic ORFがコードされている部分があります。これエクソンの上流側です。このエクソン部分には、Intron Binding Sequence II ,Intron Binding Sequence Iと呼ばれる配列があります。このIntron Binding Sequence II配列と相補的な配列を持っていて, 水素結合を形成できるような配列が 1番目のアームの中にはあります 。このような配列はExon Binding Sequence IIと呼ばれます。
またエクソンのIntron Binding Sequence I配列と相補的な配列を持って水素結合を形成できるような配列が 同じく1番目のアームの中にあって,それは Exon Binding Sequence I配列と呼ばれます。 Group IイントロンとGroup IIイントロン の両方に共通なことですけれども, これらのイントロンが正しく折りたたまれて, 立体構造を作った場合に, その折りたたまれたイントロン部分のRNAは リボザイムとして働きます。
多くの場合、酵素活性を持つ物質はタンパクからできていますけれども, RNAが様々な酵素活性を持つということが 近年知られるようになりました。酵素活性を持つようなRNAは,リボザイムと呼ばれます。正しく折り畳まれたイントロンは自分自身を切り出し, 残ったエクソン部分を繋げるという活性を持った リボザイムとして働きます。
一方,spliceosomeで切り出されるイントロン入れるには,このような活性がありません。このようにGroup IイントロンやGroup IIイントロンは,spliceosomeで切り出されるような真核生物の核内イントロンとは,非常に異なった特徴を持っています
6. 3種類のイントロンの特徴

ではここでもう一度3種類のイントロについて概略をまとめてみたいと思います Group Iイントロの特徴としてその立体構造を作るイントロ内にはORFがあり, homing enzymeあるいはhoming endonucleaseと呼ばれる ゲノムDNA内の特定配列を切断するようなDNA分解酵素が埋め込まれています。
生物界での分布としては真正細菌、葉緑体、ミトコンドリアのゲノム内に広く存在します。また真核生物ゲノムでは,例外的にリボソームRNA遺伝子の中に埋め込まれていることがあります。 Group IIイントロンは,Group Iイントロよりも大きくて複雑な立体構造をもち,Group Iイントロと同様に,イントロ内にORFがあります。このORF にコードされているのは 逆転写酵素やエンドヌクレースです。生物界での分布とすると,真正細菌に広く分布し,ごく稀に古細菌の中に見つかることがあります。 また葉緑体やミトコンドリアのゲノム内で多数見られます。
一方spliceosomeで切り出されるようなspliceosomal intronでは イントロン内にORFはありません。 また切り出されるイントロン部分には強い立体構造がありません 。分布としては,真核生物のゲノム内のタンパクをコードするような遺伝子の中に埋め込まれています。 これが3種類のイントロンの大まかな特徴です。
7. 生物界におけるミトコンドリア内イントロンの頻度比較

ミトコンドリアのゲノムの中には,Group IイントロンやGroup IIイントロンがあるというふうに説明しましたが, もう少し詳しく見ていきましょう。
これらのイントロンのゲノム内頻度には大きな偏りがあることがわかります。 多細胞動物については、イソギンチャクにおいてGroup Iイントロンが例外的に一つ見つかっていますが ,その他の多細胞動物のミトコンドリアにおいてGroup IイントロンもGroup IIイントロンも報告がありません。
原生動物(Protozoa)においては,Group Iイントロン、Group IIイントロンともそれほど存在頻度は多くないですが,報告をされています。 また、藻類(Algae)のミトコンドリアについても原生動物と同じで,Group IイントロンやGroup IIイントロンの報告はありますが ,非常に頻度が高いというものではありません。
対照的にカビ類(Fungi)では,Group IイントロンもGroup IIイントロンも頻繁に発見されます。
8. カビのミトコンドリア遺伝子内のイントロン頻度

これは子嚢菌のPodospora anserinaの cox1遺伝子を示しています。
ハッチ掛けのある白く示した部分,この領域1個が1個のGroup Iイントロンです。 全部で15個のGroup Iイントロンが,このcox1遺伝子内に含まれています。またGroup IIイントロンが1個あり,それをグレーで示しています。一方, この赤く示した部分はエクソンを示しています。
cox1遺伝子のエクソンの合計が1.6キロベースに対して イントロンの合計は全部で22.9キロベースもあります。この例のように,カビ類のミトコンドリアにはGroup IイントロンやGroup IIイントロンが多数発見されます。
9. Intronic ORFの翻訳
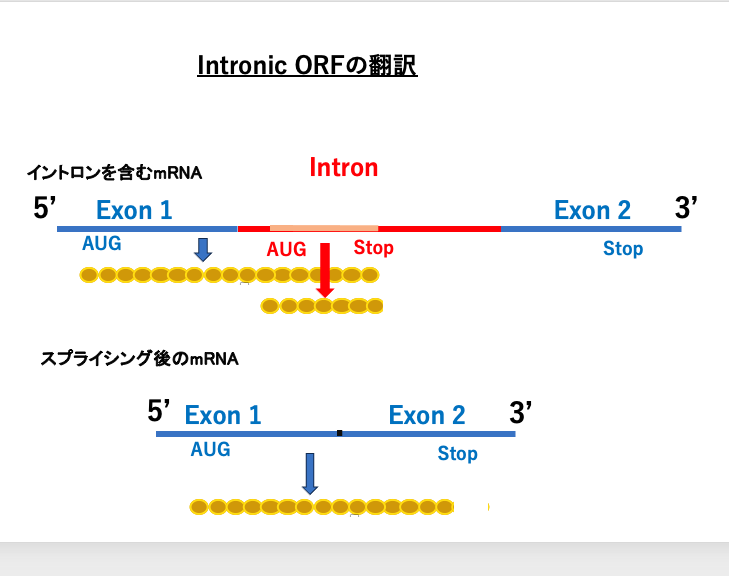
Group IやGroup IIにコードされているintronic open reading frame (ORF)が、細菌やオルガネラの細胞質でどのように翻訳されるのかを説明します。
こちらが,スプライシングを受ける前のmRNA, こちらがスプライシングが終わった後のメッセンジャーRNAを示しています。 intronic ORFが第一エキソンのすぐ下流にあり,しかも同じ翻訳フレームの場合, 第一エキソンの翻訳開始AUGコドンからintronic ORFの終始コドンまで,一続きのタンパクとして翻訳される場合があります。
あるいは,もっと単純にintronic ORFの AUGコドンから終始コドンまでが翻訳されるというやり方です。いずれの場合も,未成熟なmRNAに対して翻訳が開始されることで,intronic ORFの翻訳が実現します。
10. spliceosomeとGroup IIイントロンの切り出し反応比較
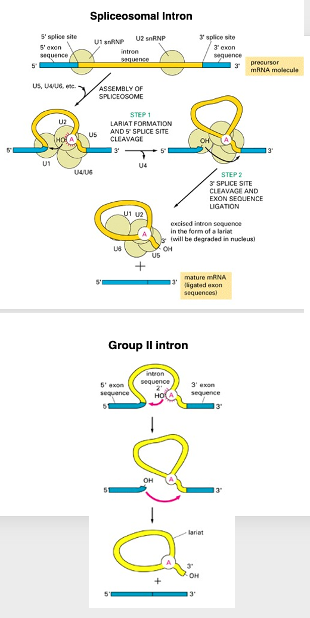
この図を使ってGroup IIイントロンとspliceosomeで切り出されるイントロンの切り出し反応を比較していきたいと思います。 こちら側がspliceosomeで切り出されるようなイントロンを示しています。 この場合、まずspliceosomeの中にあるsmall nuclear RNAが特定の場所に結合します。 例えば、U1 small nuclear RNAは上流のexon/intron境界に付着しますし、U2 small nuclear RNAはイントロン内にある特定のアデノシンの位置に付着することがわかっています。 spliceosomeがmRNAの要所に正しく結合すると,切り出し反応が開始されます。 イントロン内のアデノシンの2’ O-Hの酸素元素がexon/intron境界を求核反応で攻撃をします。
その結果,このexon/intron境界が切断され、投げ縄構造が形成されます。 投げ縄構造ができた後、この上流エキソンの3’ O-H末端の酸素元素は下流のexon/intron境界を攻撃して、イントロンが切り出されると同時に、上流と下流のエキソンが結合するという反応をします。
一方、こちらがGroup IIイントロンの切り出し反応です。 イントロン内にあるアデノシンの2’ OHの酸素元素が求核剤として働いてエキソン-イントロン境界を攻撃し、exon/intron境界が切断されると同時に,投げ縄構造が形成されます。 上流の3’ OHは下流のエキソン-イントロン境界を求核反応で攻撃し、2つのエクソンが結合し、同時に投げ縄構造を形成したイントロンが弾き出されていきます。
つまり、Group IIイントロンもspliceosomal intronも全く同じ化学反応で切り出されます。 違いとすると、Group IIイントロンではイントロン部分が立体構造を形成し、リボザイムとして働くのに対して、spliceosomal intronでは,そのイントロン部分は立体構造を形成せず,リボザイムとして機能しません。そのため,spliceosomeというふうな RNAの切断と結合活性を持つ装置が必須となっています。
11. Group IイントロンとGroup IIイントロンの切り出し反応比較
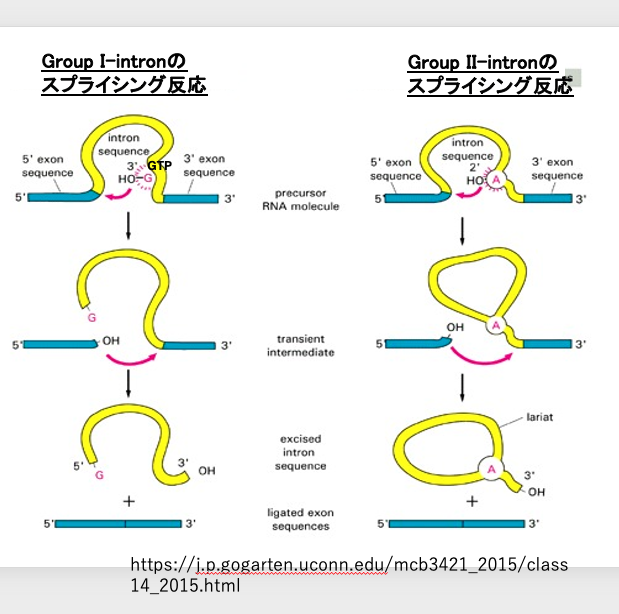
今度は,この図を使って、Group Iイントロンのスプライシング反応と、Group IIイントロンのスプライシング反応を比較していきます。 Group Iイントロンの場合、イントロンに結合しているGTPの3 prime O-Hの酸素元素が、上流のexon/intron境界を求核的に攻撃して切断します。
次に,エクソンの切断点の酸素元素は、下流のexon/intron境界を求核的に攻撃します。その結果、イントロンが切り出されて弾き出されると同時に、2つのエクソンが結合されるという化学反応が起こります。
この反応は,Group Iイントロンが立体構造を形成することで生じたribozyme活性により進行します。 このようにGroup Iイントロンの切り出し反応は、Group IIイントロンやspliceosomal intronの切り出し反応とは違っています。
Spliceosome内にあるsmall nuclear RNAの立体構造とGroup IIイントロンが形成する
立体構造の類似性
12. spliceosomeの外観
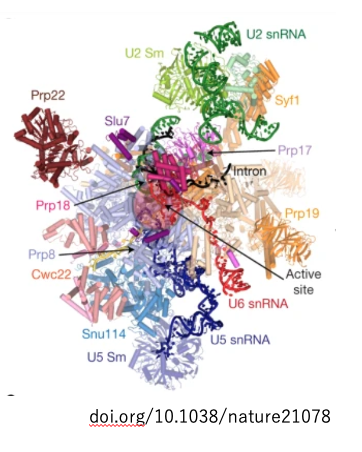
Spliceosomeは,U1、U2、U4、U5、U6と呼ばれる5種類のsmall RNAと50から100種類ぐらいのタンパクの複合体です。
Small RNAの長さは,150塩基程度で,ウラシル塩基が多く含まれているので,Uという頭文字が付けられています。 タンパクの数は生物種によって異なります。
13. Group II intronが形成する立体構造
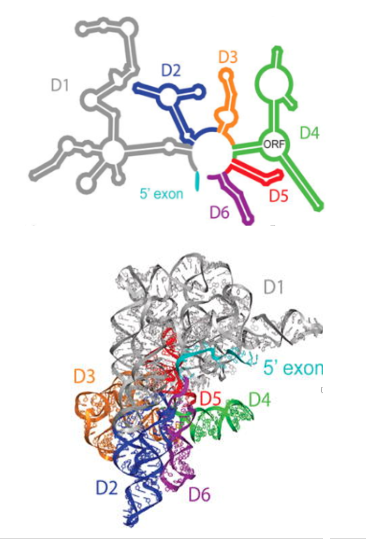
上図がGroup 2イントロンRNAの二次元の模式図です。
下の部は,立体図です。spliceosomeが切り出すイントロン部分は,大きな立体構造を持ちませんが,Group 2イントロンは,それ自体が大きくて複雑な立体構造を持っています。
ArmあるいはDomainと呼ばれる構造がD1からD6まで6つあります。D4にはOpen Reading Frameが含まれます。
14. small nuclear RNAが形成する切り出し反応の活性中心
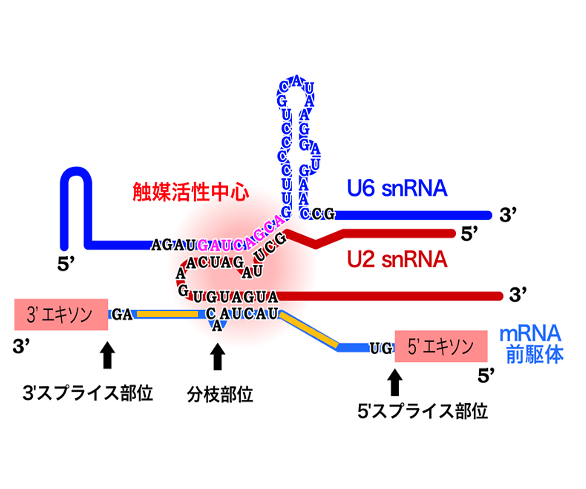
Group IIイントロンにおいて,そのRNAが正しく折りたたまれれば自らを切り出し,exonを結合する活性を持つリボザイムとして働くということをお話ししてきましたが、spliceosome の中にあるsmall nuclear RNAも正しく折りたたまれ,会合することでリボザイムとして働くということが分かっています。
左図ではU2 small nuclear RNAとU6 small nuclear RNAが会合した複合体が,切り出される投げ縄構造の結び目に位置するアデノシンに結合している場面を描いていま。spliceosome では,small nuclear RNAが複合体を形成し, メッセンジャーRNAに付着することでスプライシング反応が起こります。
spliceosomeはタンパクとRNAの複合体ですが ,スプライシング反応の活性中心はRNA側にあってタンパク質にはありません。 タンパクは主としてsmall nuclear RNAが正しい立体構造を形成し,正しく配置されるのを助けるという目的をしていると考えられています。
15. Group IIイントロンの折り畳みを介助するMaturaeタンパク
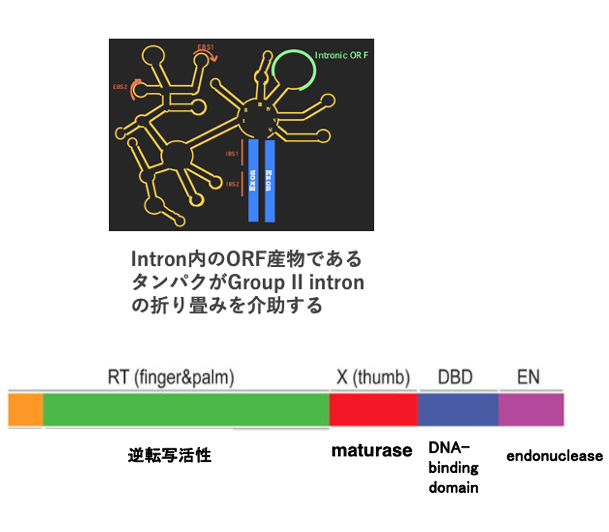
spliceosomeの中にあるsmall nuclear RNAが正しく折りたたまれるためには, タンパクの介助が必要だということをお話ししましたけれども, そのことはGroup IIイントロンの立体構造についても同じです。
そのために使用されるタンパクは,intronic ORFの産物が使われます。この図は,Group IIイントロンのintronic ORF内 の第IV-Armにコードされているタンパク質の活性を示したものです。
このタンパクは,領域毎に異なる何種類かの活性を持っています。その一つの活性が,X–domain にコードされているMaturaseと呼ばれる活性です。 Maturaseという名前ですから,何かを成熟させる為の活性を持っています。Maturaseは,intronic RNAの立体構造を正しいものに成熟させる活性を持っています。自分自身の立体構造の成熟に必要なタンパクを,自分自身のintronic ORF内に持っています。
16. 切り出し反応を担うribozyme部の立体構造が類似
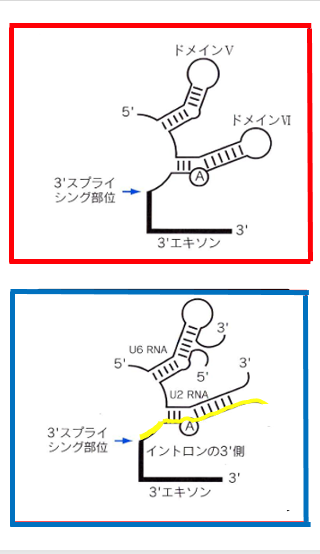
Spliceosomal イントロンとGroup IIイントロンの切り出しの化学反応は全く同一でした 。このように非常に類似した性質を持つ2種類のイントロンなのですが, 更に,intronic RNAの立体構造についても相似性があるということがわかってきました。
この図では,Group IIイントロンの中のDomain-5とDomain-6を示しています。Domain-6で,投げ縄構造の結び目に相当するアデニン塩基がここにあります。他方,こちらは,spliceosomeにおいて, U6- small nuclear RNAとU2 – small nuclear RNAが複合体を作り,切り出すべきmRNAのexon/intron境界に結合した様子を示しています。
投げ縄構造の結び目に相当するアデニン塩基がここにあります。両者において,切り出し反応を担うribozymeの立体構造は非常によく似ていることがわかるかと思います。
Group IIイントロンを起源としたSpliceosomal intoronの誕生
17. 真正細菌の遺伝子転移に伴うイントロンの古細菌ゲノムへの移行
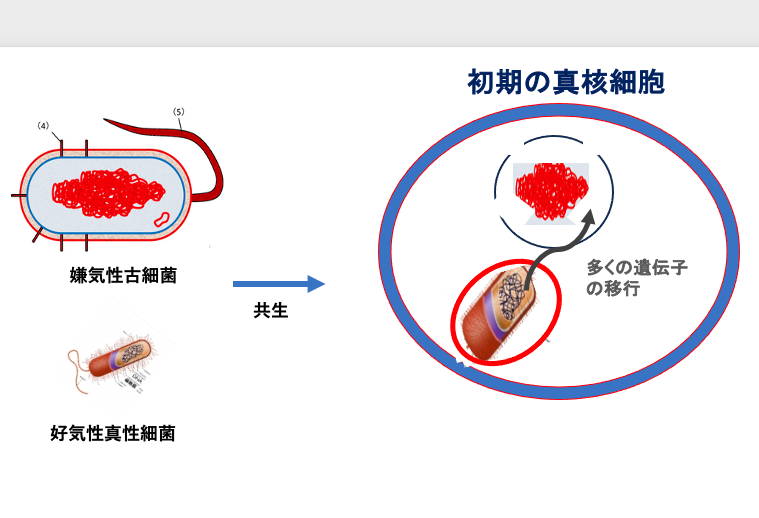
Spliceosomal intronはGroup 2 intronを起源としているという説について説明をしていきます。
真核生物は古細菌が真正細菌を飲み込み,共生させることで誕生しました。現在,古細菌にはGroup IIイントロンが ごく稀に見られるだけで Group Iイントロンは見つかっていません。 一方,古細菌に飲み込まれた真正細菌には Group IIイントロンや Group Iイントロンが多く,発見されています。
誕生初期の真核生物においては,飲み込まれた方の真正細菌の遺伝子の多くは,古細菌の核ゲノムに移行したことが知られています。このような遺伝子の転移は,当然, Group Iイントロンや Group IIイントロンの移行も伴うことになります。
古細菌の核ゲノムに移行した Group IIイントロンが, やがて現在見られるような Spliceosomal intronに進化したというふうに考えれば, Group IIイントロンの生物界内での分布や, 2つのイントロンの切り出し機構の類似性を,うまく理解することができます。
真正細菌からGroup Iイントロンも, 先ほどお話しましたように 古細菌の核に移行したと思われますが,現在Group Iイントロンは 真核生物のリボソーム遺伝子の中にしか 見られません。 この理由については,うまい説明が見つかっていません。
18. イントロンを持たない相同遺伝子へのGroup IIイントロンの挿入:Retrohoming
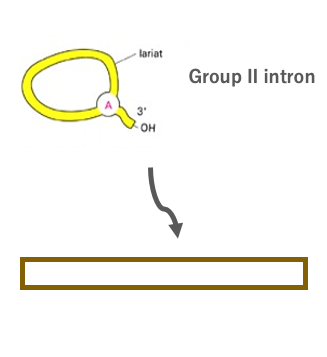
次に切り出されたGroup IIイントロンが,イントロンの切り出しの逆反応により,ゲノム内の特定の塩基配列を持つ部位に挿入されるという,イントロンの転移反応について解説をしていきます。
多くの場合,そのような特定の塩基配列は,Group IIイントロンを持つ遺伝子の相同遺伝子であって,イントロンを含まない遺伝子中に見出されます。このようなGroup IIイントロンの転移反応はRetrohomingと呼ばれます。
切り出されたspliceosomal intronも,同様の投げ縄構造のRNAですが,Retrohomingはspliceosomal intronでは通常起こらない反応す。
19. Retrohoming: 逆splicing反応とそれに続く逆転写反応
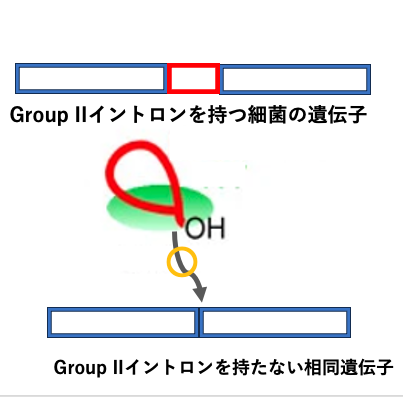
Group II intronのretrohoming機構について説明をしていきます。こちらの図がグGroup II intronが挿入されている遺伝子で,こちらは,その相同遺伝子ですが,イントロンを持っていません。
まずイントロンを持つ遺伝子からmRNAが作られ ,その未成熟なmRNAからGroup II intronが切り出されます。切り出されたイントロンは,イントロン持たない遺伝子の ,イントロンを持つ遺伝子で言えば,そのexon/intron境界に,イントロンの切り出しの逆反応で挿入されていきます。
新たにイントロンが挿入される位置が,もともとイントロンがある位置と同じなのでhomingと呼ばれます。また,この反応には逆転写酵素が関与しますので,レトロという接頭語が付加されて,retrohomingと呼ばれます。
20. Group II イントロンのRetrohoming反応
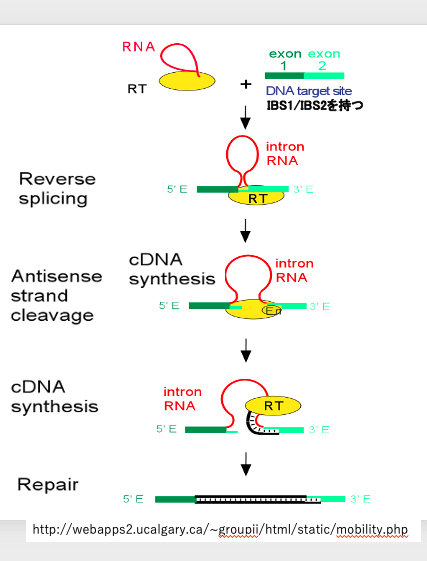
Group II intronのretrohoming反応について 詳しく説明をしていきます。 これは,投げ縄構造を持っている 切り出されたGroup II intronです。 Group II intronの切り出しに必要な タンパクはまだ付着したままです。 このタンパクの中には逆転写酵素活性なども 含まれています 。
ここに書いてあるのがGroup II intronイントロンの1番目のアームの中にある,IBS-Iと IBS-IIが連結して形成されるのと同じ配列を持つ DNAで,その配列がreverse-splicing反応における標的配列となります(このIBS-1と IBS-IIについても 後でもう一度説明をします)。
この標的DNA配列に対してreverse-splicing,つまりイントロンの切り出し反応と 全く逆の反応 により,intronic RNAの挿入反応が起こります 。この反応では,まず標的DNA鎖のうち 一本のDNA鎖にintronic RNAが結合します 。
次に,もう一方のDNA鎖がendo-nucleaseによって 切断されます。切断された DNAの3’末端から 逆転写酵素によるcDNA合成が始まります。 初めのcDNA合成が完了すると,intronic RNAは分解され,DNA鎖に生じたgap部分が埋められることにより,intronic RNA配列は最終的には 2本鎖DNA化されます。これがGroup II intronのretrohoming反応です。
21. Group IIイントロンが挿入されるゲノム内の標的配列
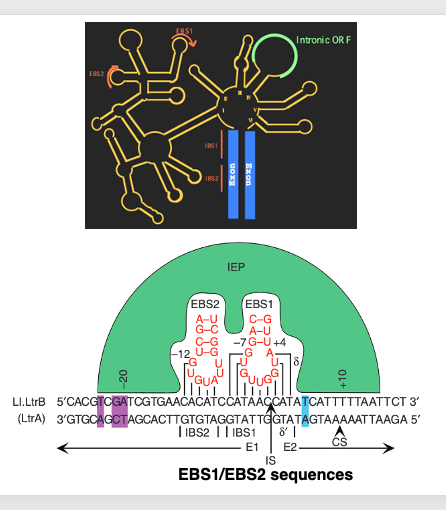
上図はGroup IIイントロン含んでいる未成熟なメッセンジャーRNAですね。 上流に位置するエキソン部には,IBS1(上図の赤文字部分)とIBS2が連結したRNA配列があります。 一方,イントロン部分にはIBS1と相補的な配列を持つEBS1, IBS2と相補的な配列を持つEBS2があります 。Group IIイントロンの切り出しには,これらのRNA配列間で安定な水素結合が形成されることが重要です。,BS1とEBS1、IBS2とEBS2の間での水素結合形成です。
それに対して,Reverse-splicing反応においては,切り出されたGroup IIイントロンのRNA配列であるEBS1とEBS2は,それぞれIBS1、IBS2と相同なDNA配列と水素結合を形成する必要があります。 splicing反応では,exon/intron境界を決定するのが,RNA-RNA間の水素結合だったのが,reverse-splicingにおいて,イントロンの挿入位置を決定しているのは,RNA-DNA間の水素結合ということです。
22. Intronic ORFにコードされれいる酵素活性
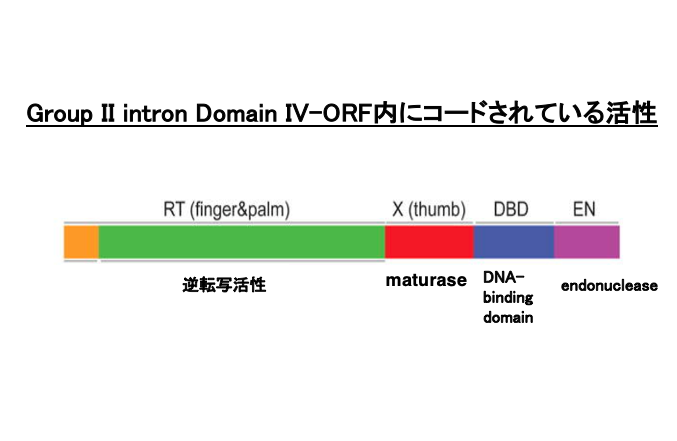
Group II intronのreverse-splicingには 逆転写酵素活性とエンドヌークレース活性などが必要とされます。 ではそういう酵素活性がどこにコーディングされているのかということについて解説をします。
これはGroup II intronのDomain-IVにコードされているintronic ORFにコードされたタンパクの酵素活性を示しています 。この一つのタンパクの中に逆転写酵素活性と エンドヌークレース活性とDNA binding活性がコードされています。 イントロンのスプライシング前にintronic ORFが翻訳されて, つくられたタンパクがsplicingにもreverse-splicingにも利用されています。
23. ゲノムに埋め込まれた利己的な遺伝的因子:Group II intron, LINE
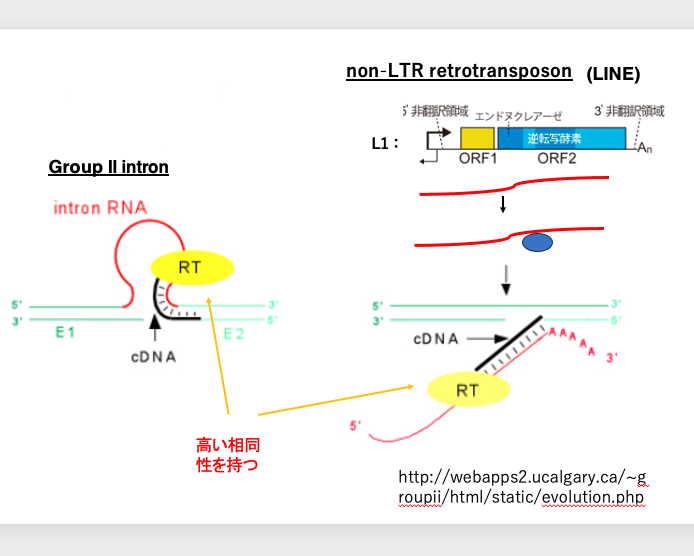
Group II intronはreverse-splicingによってイントロンをコードするDNA領域を増やすという点において,selfish genetic elementですが, ゲノムの中には,同様なメカニズムで自分自身をコードするDNA領域を増やしていくようなselfish genetic elementがゲノムの中にはありますので紹介します。
その一つが,Non-Long terminal repeat Retrotransposonに分類されるLINEです。 これはヒトゲノム内にあるLINEの一種,L1を示しています。 L1の内部には,ORFがあり,そこにはendonucleaseと逆転写酵素がコードされています 。ORFから生成されたタンパクが作られは,そのタンパクをコードしていた mRNAに付着して, 複合体を作ります 。この複合体はゲノムDNAをランダムに攻撃していきます。 エンドヌークレースがまず2本鎖DNAのうち1本鎖を切断し,その切断点からLINEのメッセンジャーRNAを鋳型とした逆転写が始まります。最終的には,LINEのメッセンジャーRNA は,2本鎖化されてゲノムDNAに埋め極まれます 。
このようにしてLINEという分子はゲノム内のランダムな場所に自分自身のコピーを増やしていくことができます。 面白いことにこのLINEが持つ逆転写酵素は,Group II intronが持つ逆転写酵素と高い相同性を持つことがでは知られています。自己のmRNAを逆転写して2本鎖DNA化する能力を持つ分子は,selfish genetic elementとしての性質を持っています。
24. Homing : Group I イントロンのintron-less 相同遺伝子への侵入
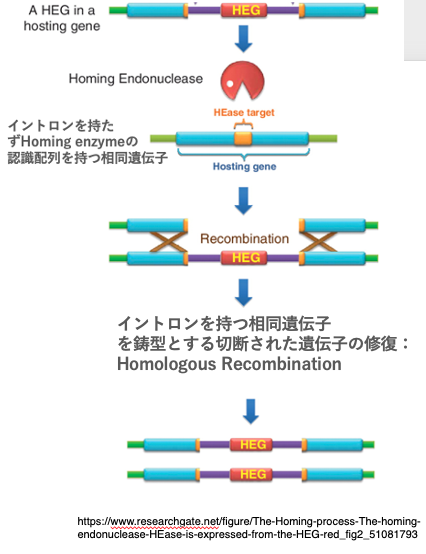
次にGroup I intronのhomingについて解説をします。Group I intronもhomingと呼ばれる反応を通して ,遺伝子の特定なサイトに自分自身をコードするDNA断片を挿入し, そのコピーを増やしていくことができるselfish genetic elementであるというふうに考えられています。 ここに示した遺伝子はGroup I intronを持ち そのイントロン内部にはintronic ORFとしてhoming enzyme (HE)あるいはhoming endonuclease と呼ばれる酵素活性がコードされています(図中:HEG)。 HEは I5から20塩基という非常に長いDNA配列を認識して,その配列の中央部付近で2本鎖DNAを切断するような酵素です。この図に示されたHEの認識配列は, このイントロンが挿入されている上流エキソン部と下流のエキソン部分 ,この2つのエキソン部分の配列が連結された配列が認識配列です。
つまり, このGroup I intronをコードするような遺伝子に対して,このGroup I intronをコードするDNA領域が抜けたintron-lessの遺伝子では,その内部にHEの認識配列を持つことになります。一方,Group I intronが挿入されているこの遺伝子では,認識配列がintronic DNA配列により,分断されています。
このイントロンを持たない遺伝子は,HEに出会うと,認識配列を持つが故に2本差DNAが切断されてしまいます。2本鎖DNAがHEによって切断された場合,それは直ちに修復される必要性があります。 しかし,切断点をそのまま連結したのでは再び HEによって認識される配列となってしまいますから切られてしまいます。 HEによる切断を免れて,なおかつ 正常な遺伝子の機能を保持できる方法が一つだけあります。
それはイントロンを持っている相同遺伝子を鋳型として,組み換え修復を行うことです。ここに示したのが,切断された遺伝子ですね。 一方,これはグGroup I intronを持っている相同遺伝子です。イントロンを持たない遺伝子が切断されたら,イントロンを持つ相同遺伝子との間で, 組み換え反応を行い,intronic DNAを内部に取り込んだ遺伝子となることで,HEによる切断を免れることができます 。しかも,取り込んだのはintronic DNA fragmentなので,メッセンジャー RNAでは,その部分は 取り除かれてしまいます。
したがって生産されるタンパクは正常な機能を持っています。このように,HEを持っているような遺伝子とintron-lessの相同遺伝子が ,同じ細胞内にある場合, intron-lessの相同遺伝子は, Group I intronをコードするDNA領域を 取り込むことによってのみ,HEの切断から免れることができ, なおかつ正常なタンパクを生産することができます。 intron-lessの相同遺伝子が,相同組み換えによりイントロンを持つ遺伝子になるような反応はHomingと呼ばれます。この反応には逆転写酵素は関与しないので,retro-homing反応ではなくて homing反応と呼ばれます。
25. 遺伝子工学のToolとしてのHoming Enzyme

このように長い認識配列を持つhoming enzymeは,現在では遺伝子工学 のツールとして利用されています。 ここに示したのはI-SECIという名前 が付けられているホーミングエンザイム の一種です. このホーミングエンザイムの認識配列は18塩基長です。この酵素は,酵母の一種である Saccharomyces cerevisiaeのミトコンドリア のGroup I intronのORF にコードされています。
認識配列が18塩基長です。4の18乗というは68億ですから,単純な確率計算では,このような配列は68億塩基長に1回しか現れないことになります。ヒトゲノムが30億塩基長であることを考えれば,非常に特異性の高い酵素という事がわかります。それゆえに,ゲノムを非常に大きく断片化したい場合などに,使用されています。
26. Intronを持つ遺伝子とIntron-lessが出会う環境:Bacterial conjugation, Fusion of Mitochondria and Plastids
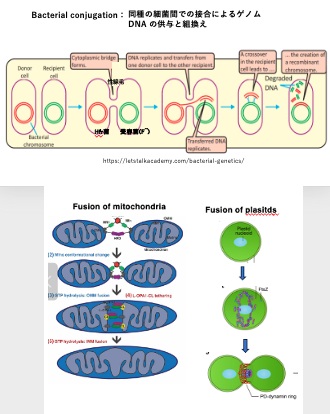
Group II intronのretro-homing、Group I introのhomingについては, イントロンを持っている遺伝子とintron-lessの相同遺伝子が ,同一細胞内で共存するという状況が必須です。しかし,多くの細菌では,各遺伝子は1コピーのみが存在していて,イントロンを持つ遺伝子とintron-less遺伝子が同時にゲノム内にコードされているような状況ではありません。では,どのようにして,イントロンを持っている遺伝子とintron-lessの相同遺伝子が共存する状況が出現するのかということについて解説をしていきます 。
バクテリアではconjugationが知られています 。conjugationというのは,同種の細菌間での接合による ゲノムDNAの共有と組み換えを指します。バクテリアでも,性が存在していて,雄のバクテリアから雌のバクテリアにDNAが供給されることがあります 。雌のバクテリアとメスのバクテリアがpilusを通じてつながります。 性線毛を通して,雄のバクテリアから雌のバクテリアにDNAの共有が始まります。 このようにしてメスのバクテリアの中で オスのバクテリアが持っていたイントロンレスの遺伝子と メスのバクテリアが持っているイントロンを含む遺伝子が 同一細胞内に共存するという状態が生まれる場合があります 。
また真核生物では受精・接合を通じて核同士が融合しますが ,同時にミトコンドレア同士・葉緑体同士も融合します。 その時にオスの持っているミトコンドリアDNAの遺伝子がintron-lessで ,メスの持っているミトコンドリアDNAの遺伝子がイントロンを含む場合, intron-lessの遺伝子とイントロンを持っている遺伝子が 共存するというふうなことが起こります。そういうふうな状況下では,retro-homing やhomingが起こります。
これでイントロン進化についての解説を終わります。
コメント