この講義の内容は下記のYouTubeサイト(再生リスト:生命科学を専攻する学生の為の分子生物学講義)で
動画として視聴できます
https://youtu.be/trYITxR5SgM
1. 講義内容
- 真核生物にのみ存在するRNA silencing の起源
- RNA interferenc:siRNAが関与する転写後発現抑制
- 長い完全相補配列を持つRNA鎖からのduplex siRNAの生成
- Passenger strandの切断とAgoからの放出
- Guide strand選択の原理
- siRNAのseed配列による標的mRNAのスキャン検索
- RISC結合による標的mRNAの切断または翻訳阻害
- 2次的なsiRNAを生産するメカニズム
- miRNAによる転写後発現抑制の機構
- miRNAによる転写後発現抑制の経路
- siRNAとmiRNAによる転写後翻訳抑制の比較
- 植物ウイルスにおけるRNA silencing suppressor
- piRNAによる転写後発現抑制の機構
- piRNA とmiRNAの比較
- piRNAクラスターの転写物の切断と活性化Piwi-piRISC の生成経路
- Dual-strand clusterの転写物から成熟piRNAが生産される経路
Key words: RNA interference, siRNA, miRNA, piRNA, RNA silencing, Argonaute, RNA virus, RISC, TRBP, Guide strand, Passenger strand, 2次的なsiRNA, 生殖細胞, トランスポゾン, uni-strand piRNA cluster
2. RNA silencingの2経路
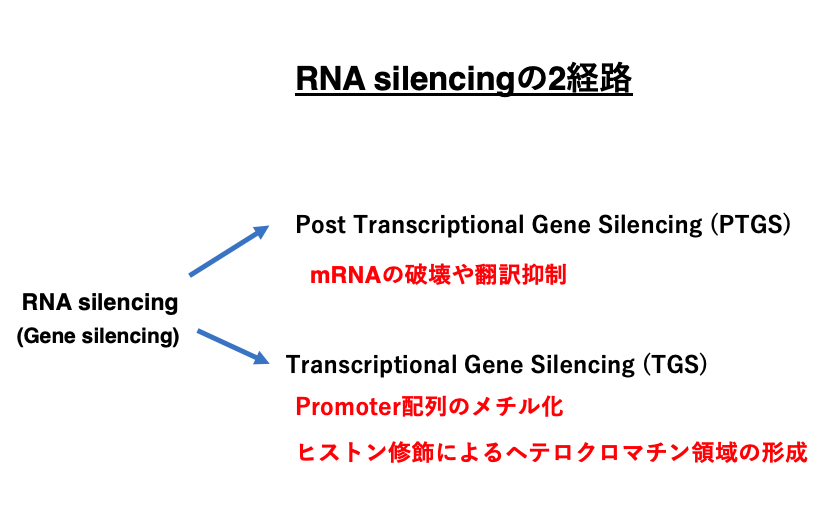
siRNAや miRNA、piRNAなどのnon-coding small RNAはmRNAからタンパクが合成されるのを妨害するPost Transcriptional Gene Silencingの他に,Promoter配列のメチル化やヒストン修飾を介したヘテロクロマチン領域の形成による転写抑制も誘導することが知られています。
このようにRNA silencingには2経路がありますが, Small RNAによる転写抑制は,この講義には含まれていません。
3. 真核生物にのみ存在するRNA silencing の起源
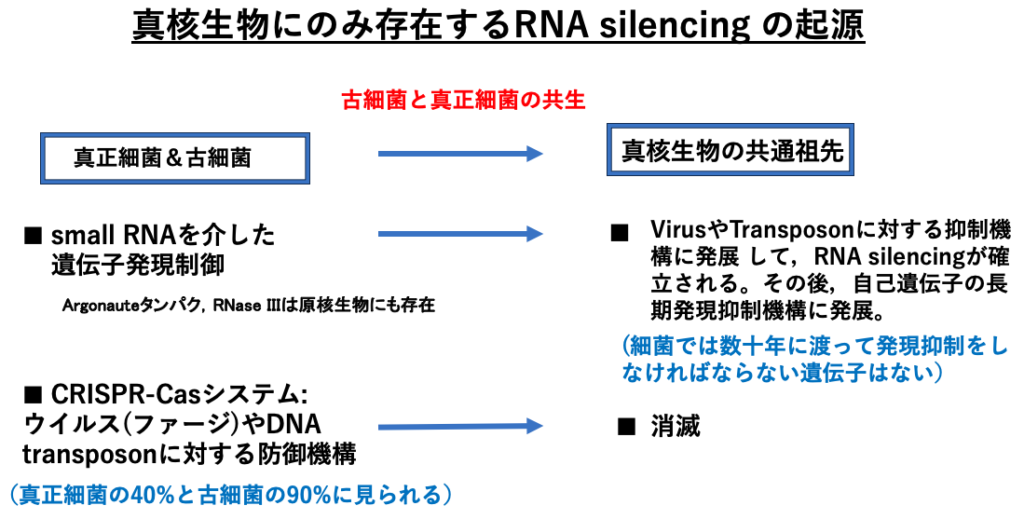
Small RNA分子によるRNA silencingは,真核生物のみが持つメカニズムで真正細菌や古細菌にこの機構はありません。真核細胞は古細菌と真正細菌の共生から生じたhybridな細胞なので,真核生物のみに存在するとされるメカニズムの多くは,実は細菌に存在していた機構が真核細胞で変化して発展したものだったという事が多くあります。
そのような視点でRNA silencing反応を見ると,原核生物においてsmall RNAによって行われていた遺伝子の発現制御が,初期の真核生物においてVirusやTransposonに対する抑制機構に発展し,真核多細胞生物ではそれがさらに発展して自己遺伝子の長期発現抑制にも用いられるように進化してきたメカニズムだと考えられます。実際にRNA interference反応において重要な役割を果たすArgonauteタンパクやRNase IIIは,原核生物にも存在しています。
細菌はウイルスや内在性のDNA transposonに対抗するシステムとしては,CRISPR-CasシステムというDNAレベルでの対抗手段を持っています。一方,RNA silencingが発達した真核生物では,この機構は失われてしまっています。
4. RNA silencingの真核生物内での広がり
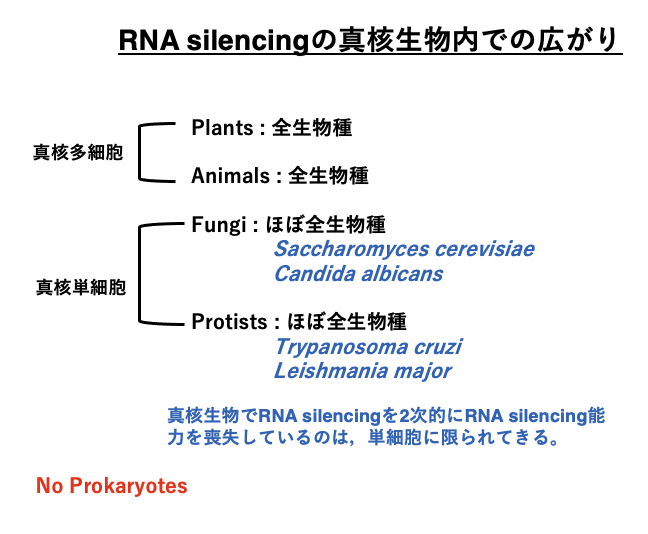
RNA silencingは,植物,動物を問わず全ての多細胞生物で見られます。真核単細胞生物であるカビ類や原生動物においても,ほぼ全ての生物種でみられますが,これらの真核単細胞生物ではこの反応を2次的に喪失したと考えられる生物種があります。例えば,出芽酵母のSaccharomyces cerevisiaeではRNA silencing反応が欠損しています。
5. siRNAの前駆体となる長い相補配列を持つ2本鎖

RNA silencingのなかで,KASEAA 52(4) 241-248 (2014)
short interfering RNA (siRNA)による転写後発現抑制について解説を始めます。siRNAの前駆体は長い相補配列を持つ2本鎖RNAです。これが切断されことでsiRNAが生産されます。
6. RdRpを介して増殖するRNA virusは長い2本鎖RNAを形成する
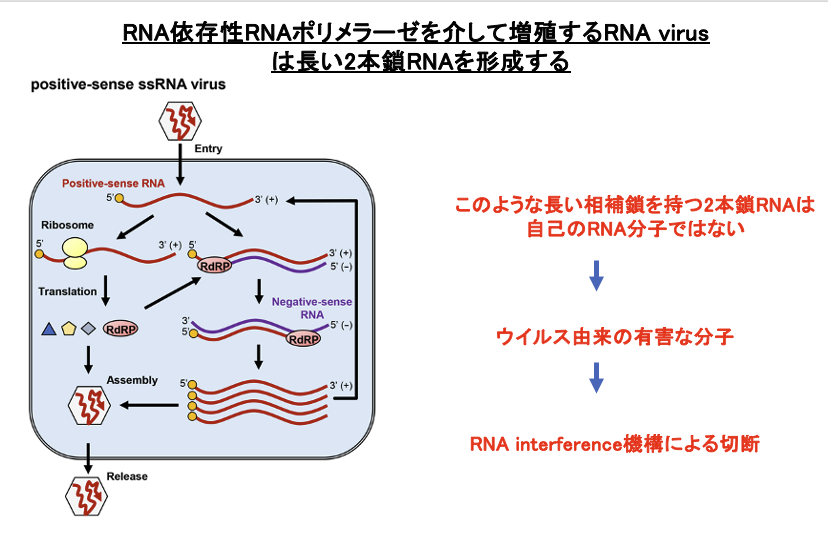
RNA interferenceの標的として,RNA virusに由来する長い完全相補の2本鎖RNAがあります。RNA virusには,逆転写酵素によりDNA分子に変換され増えるタイプのものと,RNA dependent RNA polymerase (RdRp)によって増えるタイプのものがあります。
RNA dependent RNA polymeraseによって作られるような完全相補の長いRNA分子は,真核生物の自己遺伝子の転写物には存在しません。従って,このような RNA分子を破壊することは,増殖しようとしているウイルス分子の排除機構として宿主には有益な機構だと思われます。
7. 単純化したRNAiの反応
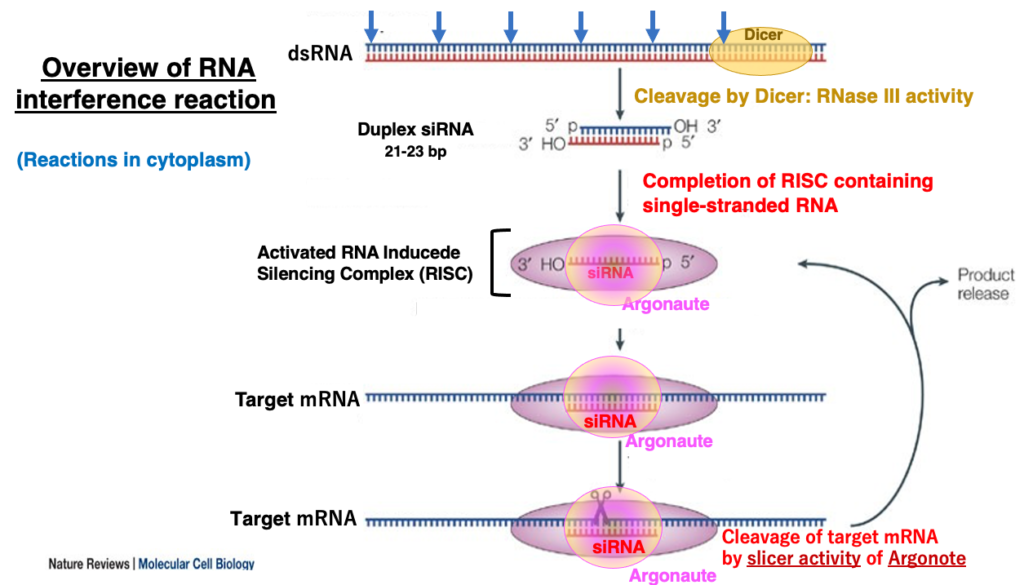
まずは,RNA interference反応の概要を説明します。Dicerと呼ばれる2本鎖RNAを特異的に切断する酵素(RNase III活性) は,細胞質の中に完全相補の長い2本鎖RNA分子があると,それを末端から21-23塩基長のsmall 2本鎖RNAにサイの目状に切断します(サイコロ=Dice)。このsmall 2本鎖RNAはshort interfering RNA RNA (siRNA)と呼ばれます。
5’-末端がモノリン酸で,3‘-OH末端が2塩基突出した形状をしています。この2本鎖のうち,一方の鎖がArgonauteタンパクを中心として形成されたRNA-Induced Silencing Complex (RISC)に引き渡されます。
2本鎖の内の一方のみが内部に残され活性化されたRISCは,内部のsiRNAと相補な配列も持つmRNAを細胞内で探索します。そのようなmRNAが見つかるとRISC内でsiRNAとmRNA間で形成され,そのduplexの中間でmRNA配列が切断されます。
このようなArgonauteが持つ切断活性はslicer活性と呼ばれます。切断が終わると,RISCは切断したmRNAから離れて,再びsiRNAと相補な配列を持つmRNAを細胞質内で探索します。
8. duplex siRNAの生成
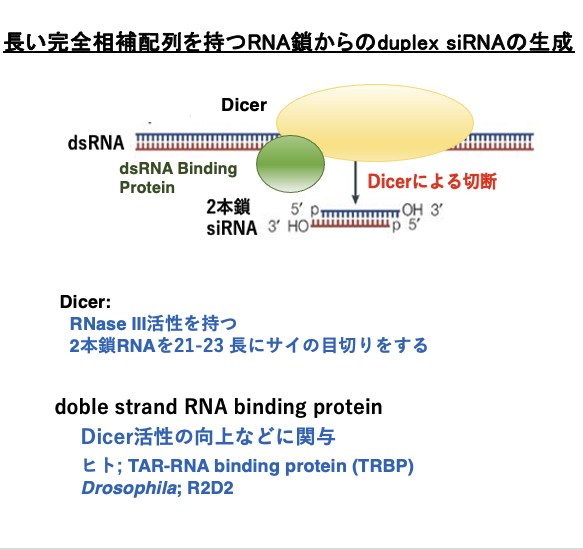
Dicerによる長い完全相補配列を持つRNA鎖の切断とduplex siRNAの生成について,もう少し詳しく見ていきます。
Dicerは2本鎖RNAを末端から21-23塩基長にブツ切りにするという,RNase III活性を持つ酵素です。Dicerはdouble strand RNA結合タンパク[ヒトの場合はTAR-RNA binding protein (TRBP)、DrosophilaではR2D2]とヘテロ複合体を形成して,2本鎖RNAを切断します。切断された短いRNA鎖は2本鎖siRNAと呼ばれます。
9. 2本鎖siRNAの特徴

2本鎖siRNAの特徴について説明をします。
2本鎖siRNAの長さは種によって多少ことなりますが,21-23塩基くらいの長さです。その3’-末端は2塩基突出の形状を持っています。また3’-末端の塩基は,2′-O-methylationのような修飾を受けている場合があります。この修飾により,siRNAの安定性や標的mRNAへの特異性が向上するものと考えられています。
2本鎖のうち,Argonaute内に残される方の鎖はGuide strand、捨てられる方はPassenger strandと呼ばれます。
10. siRNA duplexのAgoへの積み込みとpassenger strandの放出
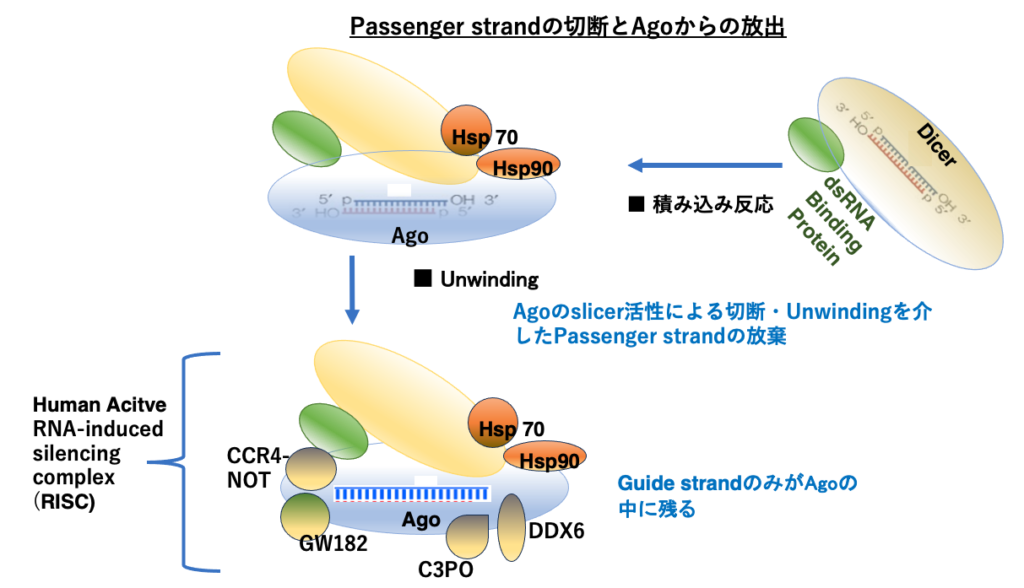
2本鎖siRNAのうちGuide strandのみがArgonaute内に残る過程をもう少し,詳しく見ていきます。
Dicerとdouble strand RNA結合タンパクの複合体により切断された2本鎖siRNAはArgonauteに引き渡される必要があります。2本鎖siRNAのうち,どちらがGuide strandとして残されるのかが考慮されてArgonauteに移されるために,Hsp70やHsp90のようなシャペロンタンパクが必要になります。
Argonaute内に特定の方向で引き渡された2本鎖RNAのうち,Passenger strandはArgonauteが持つslicer活性により切断され,Argonauteから放出されることになります。Guide strandのみがArgonaute内に残されて,さらにいつくかのタンパクが付着することで,活性型のRNA-induced silencing complex (RISC)が完成します。RISC内にあるArgonauteは内蔵するsiRNAと相補な配列を持つmRNAを見つけ出し,そのmRNAを切断することができます。
11. Dicer・dsRNA結合タンパク・Agoが会合した複合体の立体モデル
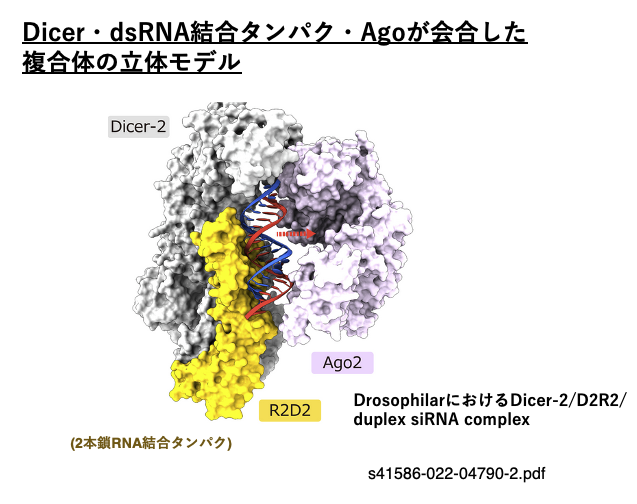
この図は,Drosophilaにおいて2本鎖siRNAがDicer-2/ 2本鎖RNA結合タンパク(R2D2)複合体からAgo-2に引き渡されようとしている様子を示しています。
12. Guide strand選択の原理
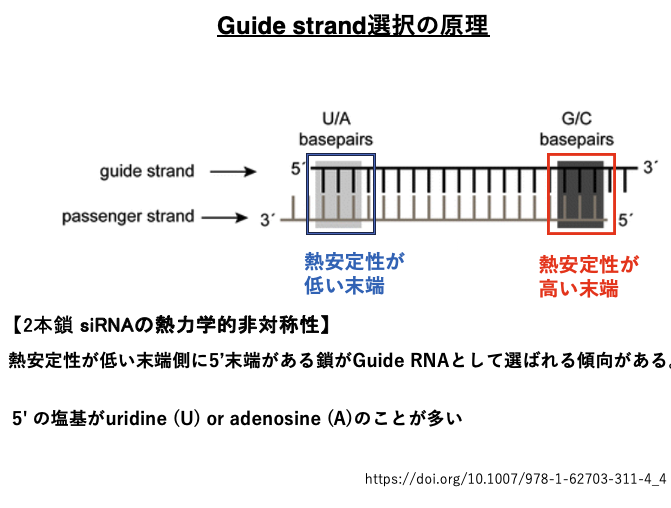
2本鎖siRNAから最終的にArgonaute内に残されるGuide strandがどのようにして決定されるのかについて解説します。
2本鎖siRNAの2つの末端付近の熱安定性(Tm)を比較します。その時,熱安定性が低い方の末端側に,5’-末端をもつ鎖がGuide strandとして選択される傾向があることが分かっています。
13. AGO のdomain配置と立体構造モデル
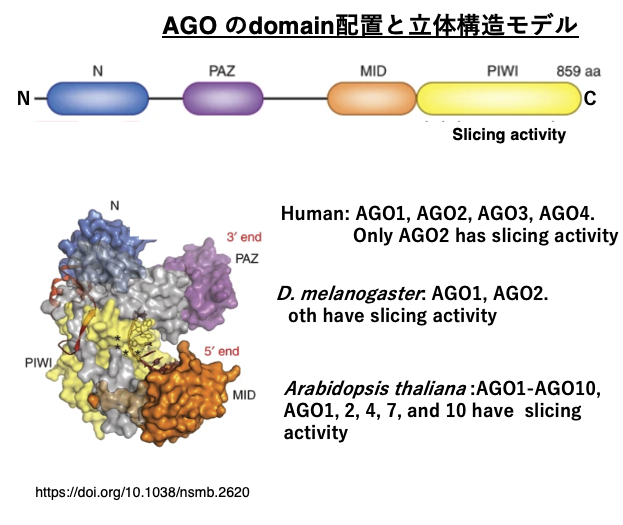
Argonauteにはいくつかのドメインがありますが,C-末端近傍にあるPIWI domain内にmRNAやGuide strandを切断するslicer活性があります。
Argonaute遺伝子はヒトでは4つ,Drosophilaでは2つ,Arabidopsisでは10個あります。生物種によっては,一部のArgonauteタンパクのみがslicer活性を持っている場合があり,slicer活性を持っていないArgonauteと役割分担をしていることが知られています。
14. Dicerで切断される長い相補配列を持つ2本鎖RNAの生成
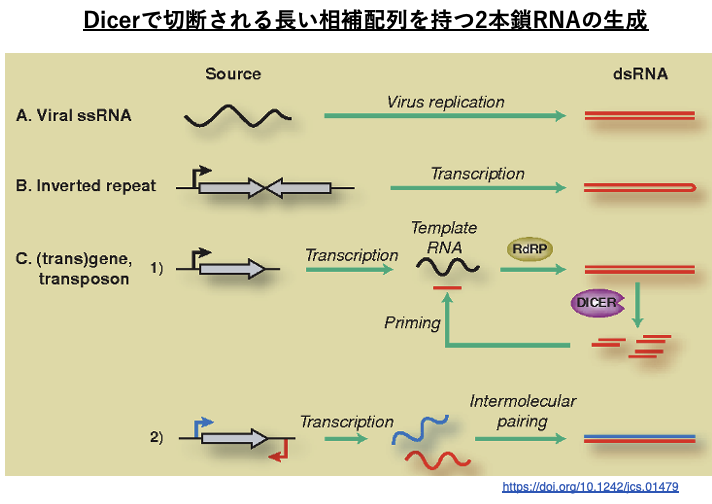
長い完全相補配列を持つRNA鎖としては,RNA virus由来の他に,Inverted Repeat構造を持つ遺伝子の転写物,遺伝子やトランスポゾンのmRNAがRNA dependent RNA polymeraseによって2本鎖化した産物,遺伝子のセンス鎖とアンチセンス鎖の両方が転写された産物などがあります。
15. 活性化したRISCによる標的mRNAの切断
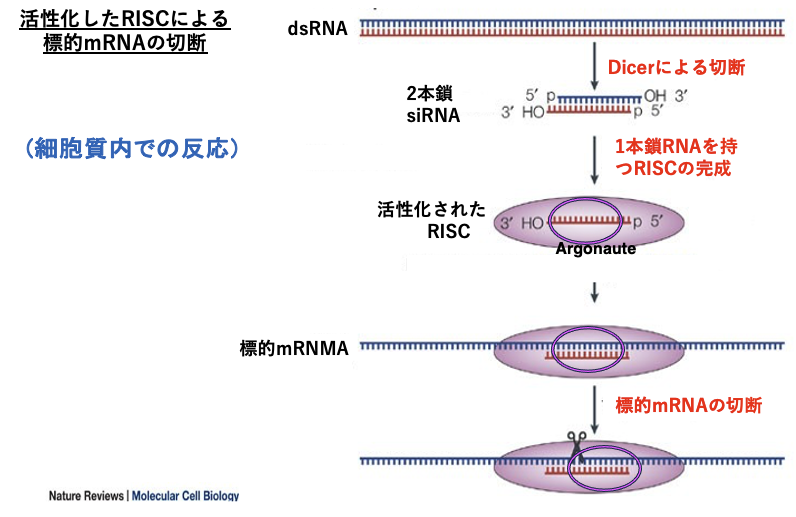
一本鎖siRNAが組み込まれて活性化したRISCが標的mRNAを探索して,切断する反応について解説していきます。
16. siRNAのシード配列による標的mRNAのスキャン検索
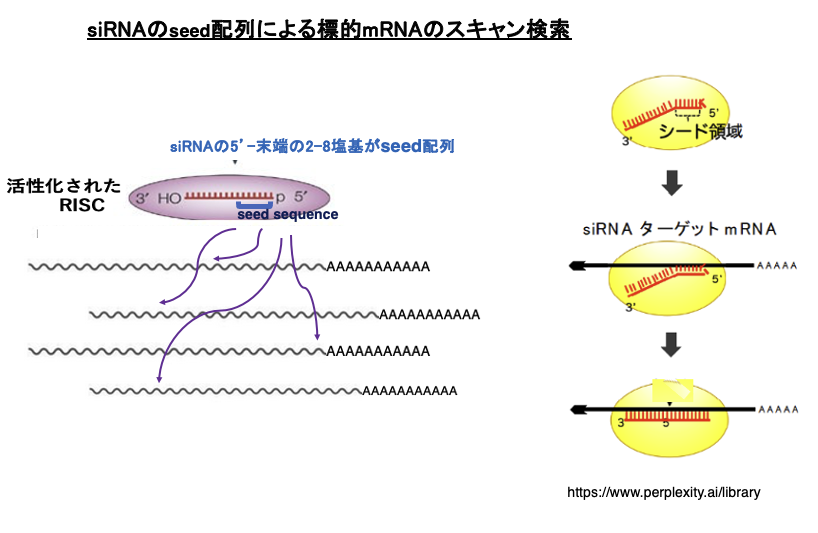
活性化したRISCによる標的mRNAの探索について解説します。siRNAの5’-末端にある2番目から8番目の塩基はseed 配列と呼ばれます。RISCはmRNAに対してこのseed 配列と相補性があるかどうかをチェックします。
その結果,相補性がなければそのタイミングでscanを中止して次のmRNAの探索に移行します。seed 配列と相補性が認められた場合のみ,相補性のチェックを継続します。その結果,完全な相補性が確認された場合,mRNAはRISC内のAgoにより切断されることになります。
17. siRNAのseed配列による標的mRNAのスキャン検索(別図)
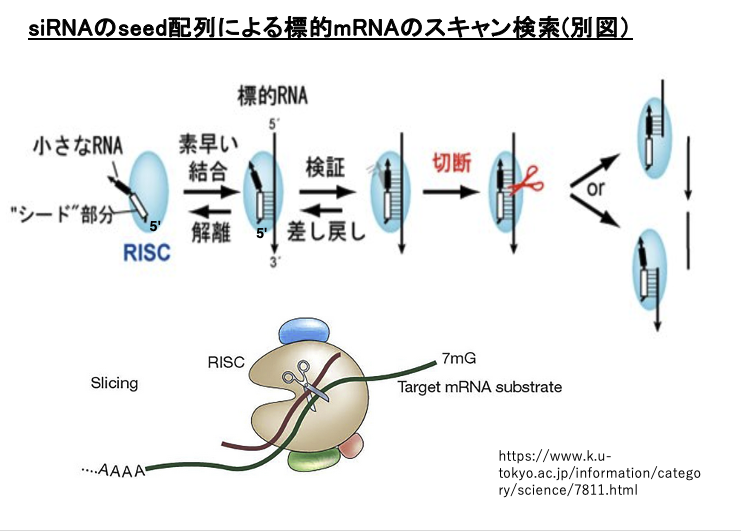
別の図でseed 配列による標的mRNAの探索を見てみましょう。siRNAの5’末端近傍のseed 配列を使って,相補性をチェックしていきます。この段階で,相補性がなければRISCはmRNAから解離していきます。相補性があれば,残りの配列に関して相補性を検証します。
18. RISC結合による標的mRNAの切断または翻訳阻害
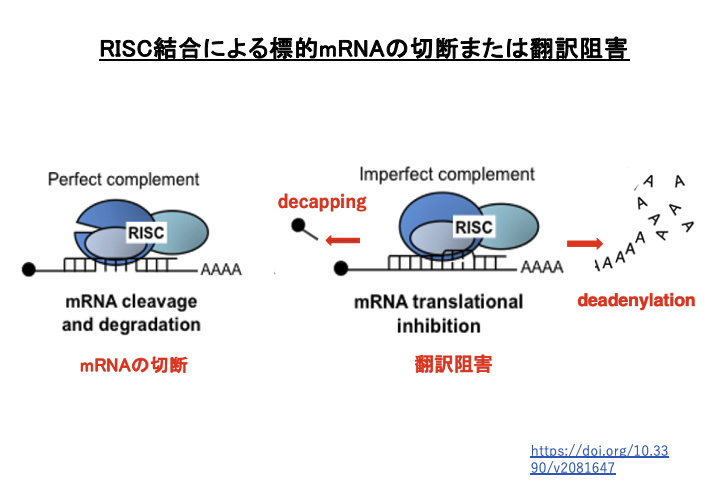
siRNAとmRNAの相補性が完全な場合は切断が起こります。一方,不完全な場合,mRNAは切断されず翻訳抑制が起こります。また,RISCに含まれているArgonauteがslicer活性を持たないタイプの場合には相補性の程度によらず,翻訳抑制が起こります。
その場合,mRNAの3’-末端のpolyA配列の分解や5’-末端にあるCap構造の除去が誘導され,mRNAの分解が促進されます。
19. 2次的なsiRNAを生産するメカニズム
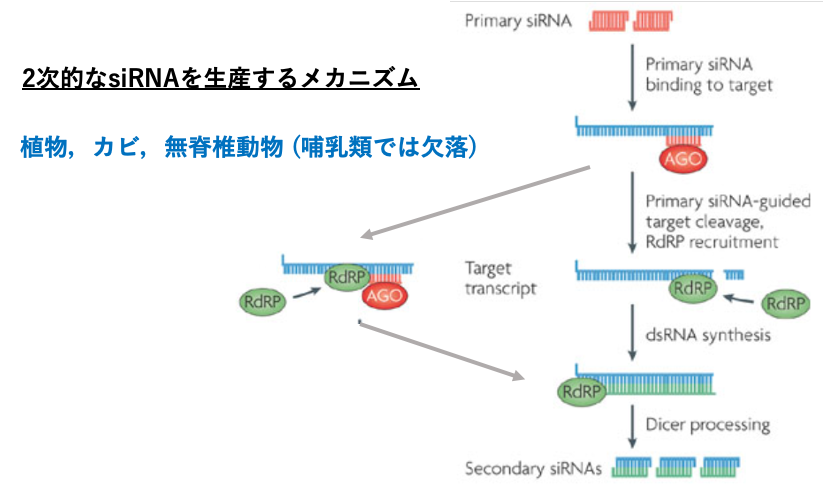
ウイルス由来の2本鎖RNAがRISCで切断されて作られた2本鎖siRNAは1次siRNAと呼ばれます。この他にsiRNAが2次的に生産される場合があります。これについて解説します。
この場合,活性化したRISCが標的mRNAに付着した後mRNAは切断されず,RNA dependent RNA polymeraseが呼び込まれて,標的mRNAが2本鎖化されます。この2本鎖RNAがDicerに切断されて生じるsiRNAは2次的 siRNAと呼ばれます。2次的 siRNAを生産する別の経路としては,RISCによって切断されたmRNAにRNA dependent RNA polymeraseが付着して,2本鎖化されたRNAがDicerで切断されて2次的 siRNAが生産されるという経路です。
このような2次的 siRNAの生産経路は植物やカビ,無脊椎動物にはありますが,哺乳類では欠損しています。哺乳類では,ウイルスからの防御手段としてRNA interferenceではなく主として免疫反応に頼っているからかもしれません。
20. miRNAによる転写後発現抑制の機構

では次にmicro RNAによる転写後発現抑制の機構について解説をします。
21. miRNAによる転写後発現抑制の経路
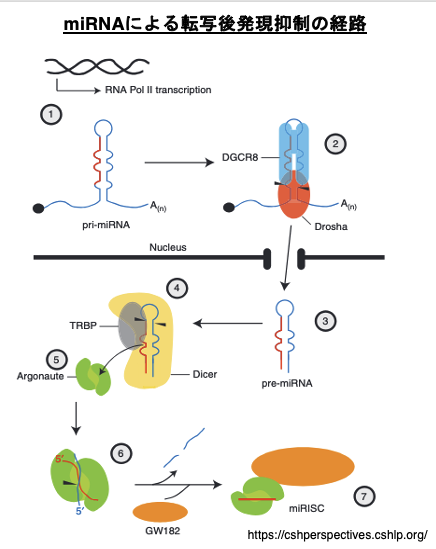
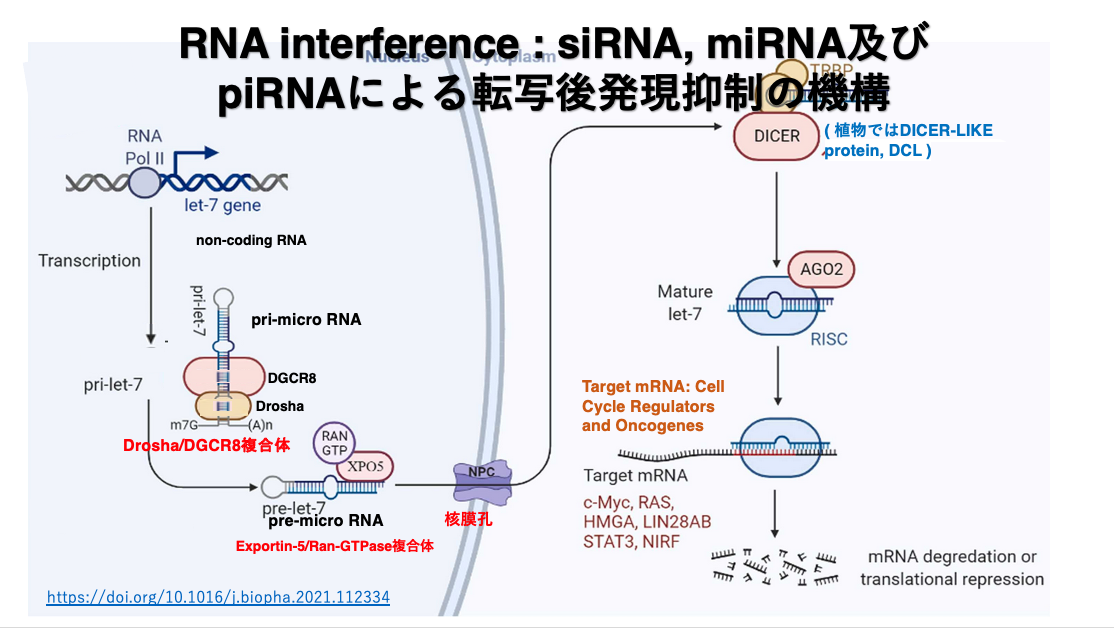
この場合,Argonauteに組み込まれるmicro RNAは,完全相補な長い2本鎖RNAからつくられるのではなく,RNA polymerase IIで転写されたヘアピン形状を持つRNAから作られます。転写されたばかりで,核内になるヘアピン形状を持つRNAをprimary micro RNA (pri-microRNA, pri-miRNA)と言います。pri-micro RNAのステム部分は,完全な相補配列ではなく一部にミスマッチが存在します。
核の中で,DGCR8がヘアピンpri-micro RNAのヘアピン構造を認識し,そこにDroshaが呼び込まれてmicroprocessor complexが作られます。Droshaは,pri-micro RNAの5’-末端や3’-末端を切り縮めます。この短くなったヘアピンRNAをpre-micro RNAと言います。precursor micro RNA (pre-micro RNA, pre-miRNA)は核から細胞質に移送されます。
細胞質の中でpre-micro RNAにDicerとTRBPが付着してpre-micro RNAからヘアピンの部分などを取り除かれて,19-25塩基の2本鎖RNAとなります。この2本鎖RNAはArgonauteタンパクに引き渡され,2本鎖RNAのうちguide strandのみが残されます。guide strandを内包したArgonauteにGW182などのタンパクが付着することで,Argonauteは活性化された状態になります。
22. ショウジョウバエの前駆体micro RNAと成熟micro RNA
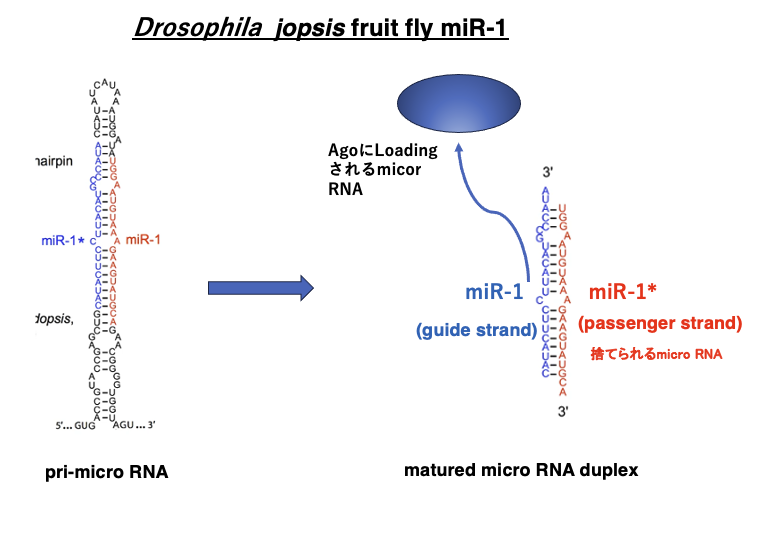
この図はDrosophilaのmicro RNA の一つmiR-1を示しています。左の図が核内にある転写直後のpri-micro RNAで,右の図が成熟した2本鎖micro RNAです。siRNAと同様に3’-末端が2塩基突出しています。
この図では,青字の配列がguide strandです。最終的にArgonaute内に残されます。2本鎖micro RNAからguide strandを選択するメカニズムはsiRNAの場合と同じで,末端配列の熱安定性の違いによって選択されます。
23. miRNAによるPost Transcriptonal Gene silensingの経路
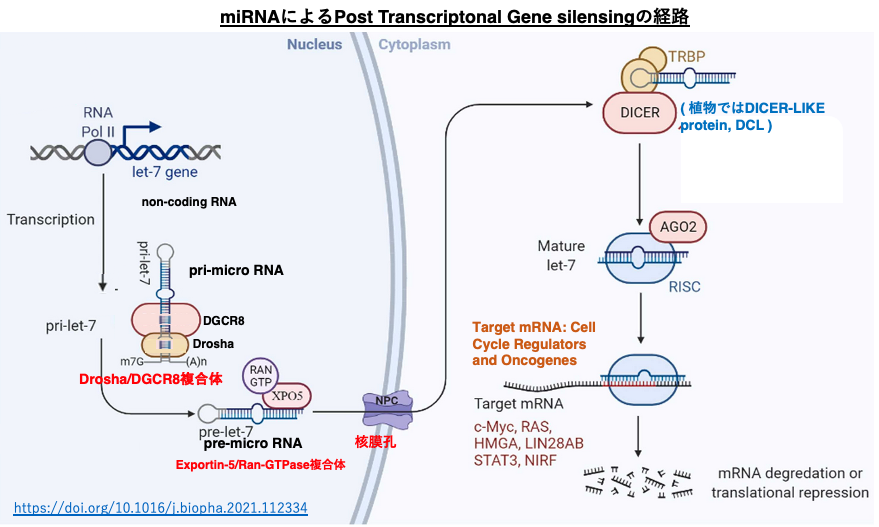
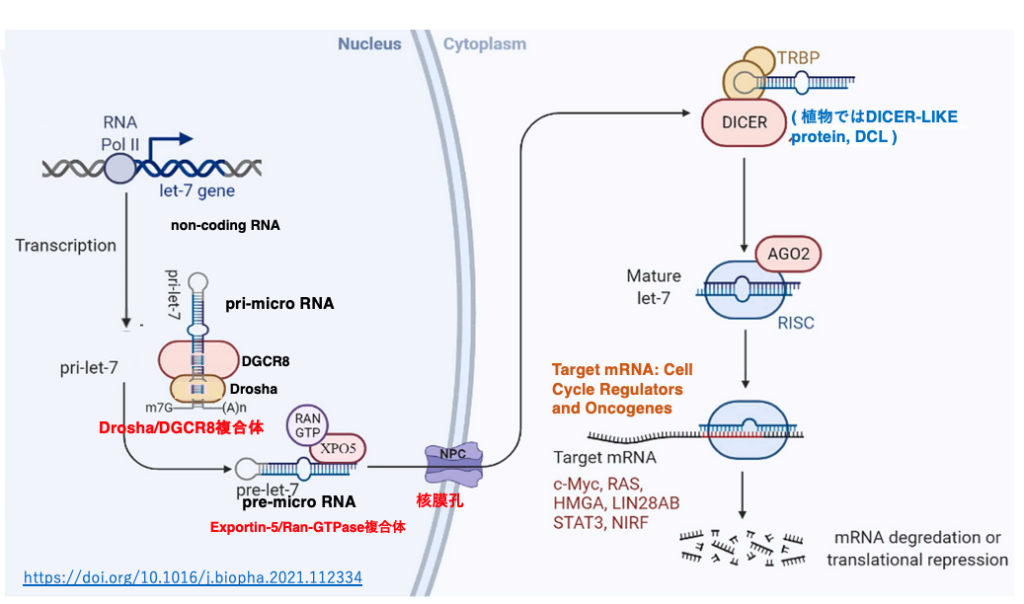
ヒトの血液性ガンの発症に関与していることが知られているlet-7 micro RNAによる転写後発現抑制について見てみましょう。
let-7遺伝子がRNA polymerase II により転写されます。転写物のpri-let-7はDroshaとDGCR8の複合体により,5’-末端と3’-末端の一部が切り取られて,pre-micro RNAとなります。これにExportin-5/Ran-GTPase複合体が付着して,核膜孔から細胞質に移送されます。細胞質内ではpre-micro RNAに2本鎖RNA結合タンパクのTRBPとDicerが付加し,22塩基長の2本鎖micro RNAに変換されます。
このRNAはArgonauteに引き渡され,guide strandのみが内部に残されます。一本鎖のguide strandとその他のタンパクの付加により活性化したRISCが形成されます。RISCは,細胞質内でguide strandと高い相補性を持つmRNAを探索します。guide strandと標的mRNAの塩基相補性は100% matchingである必要はなく,2-3塩基のmismatchが許容されます。
RISCの付加により,標的mRNAの翻訳が阻害されると同時に,分解が促進されます。これまでに,let-7 micro RNAの標的は c-MycやRAS、HMGAをコードする複数のmRNAであることが知られています。
24. siRNAとmiRNAによる転写後翻訳抑制の比較

まずsiRNAやmiRNAが作られる元のRNA分子の違いについてです。siRNAは完全な相補鎖を持つ長い2本鎖RNAから切り出されるのに対して,miRNAは,stem部分にミスマッチを持つヘアピンRNAから切り出されます。
siRNAの標的mRNAはウイルスやトランスポゾンなどの有害な因子由来であり,切断されます。一方,miRNAの標的mRNAは,細胞周期・分化制御に関する自己遺伝子由来であり,RISCの付着による翻訳阻害と末端からの分解が誘導されます。siRNAでは標的分子は一つですが,miRNAの標的mRNAは複数の場合があります。また,siRNAによる標的遺伝子の発現抑制効果はある程度強いですが,miRNAの効果はそれほど強くありません。
細胞内に人工的にsiRNAやmiRNAを導入できれば,特定の遺伝子や遺伝子群の発現を同時に抑制できる可能性があります。しかし,siRNAやmiRNAの効果の程度は生物種によって大きく異なるようです。
25. siRNAとmiRNAによる発現抑制経路の比較
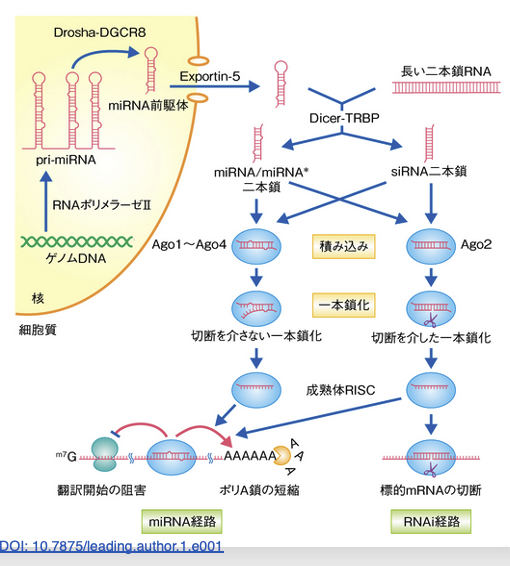
この図では,siRNAによる発現抑制の経路とmiRNAによる発現抑制の経路の経路を同時に示しています。2つの経路で,Dicer-TRBP複合体やAgo, RISCはどちらかの経路に特化しておらず,同一タンパクが両方の経路で使用されていることがわかります。
26. 植物におけるsiRNAの長距離移動
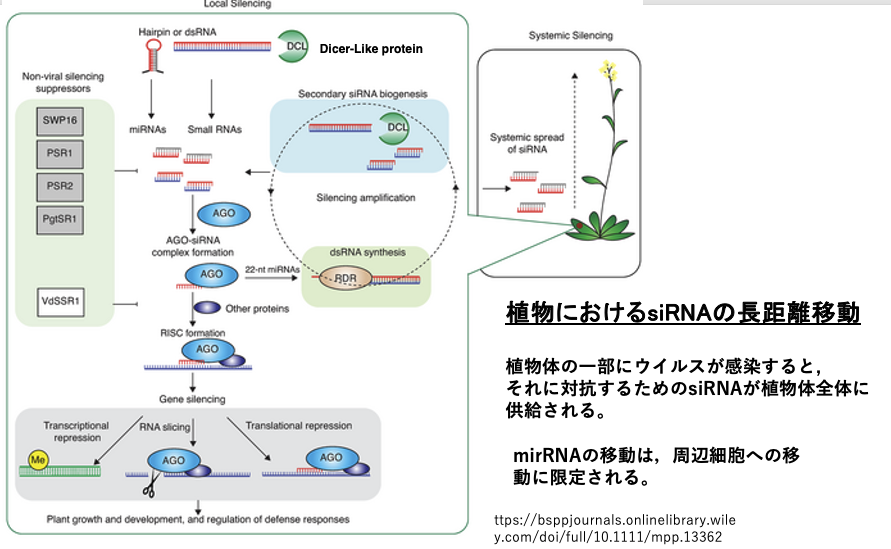
陸上植物では一部の葉にウイルスが感染してsiRNAが生産されたような場合,siRNAは師管を移動することにより植物体全体にウイルスの防御体制が広がることが知られています。一方でmiRNAの場合は,その移動は周辺細胞に広がるのみです。
植物では、通常miRNAはその標的遺伝子に対して(ほぼ)完全に相補的であり、RISCによる直接的なmRNAの切断が誘導されるのに対し、動物のmiRNAは標的となる配列がより多様である傾向があり、翻訳抑制が誘導される傾向があります。
27. 植物ウイルスにおけるRNA silencing suppressor
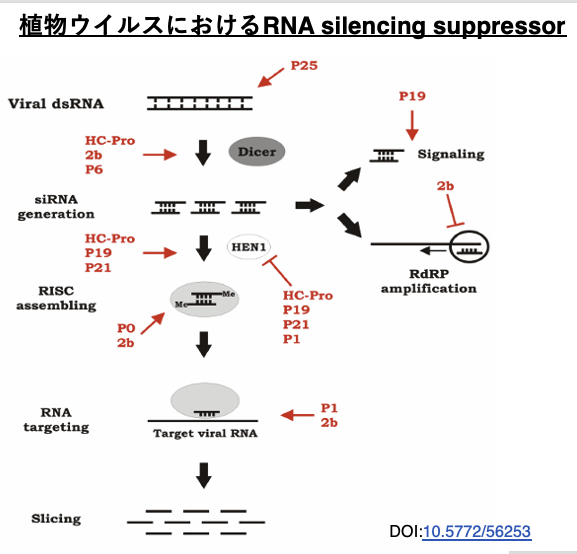
RNA interferenceは宿主が持つウイルスに対する防御装置ですが,ウイルス側も宿主のRNA interference反応を妨害するタンパクを発現させることで,これに対抗しています。
この図では,RNA interferenceの反応を妨害するウイルス側のタンパクを赤字で示しています。このように様々なRNA interferenceのステップを妨害するタンパクをウイルス側も持っており,宿主のRNA interferenceによるウイルス増殖阻害に対抗しています。
28. RNA silencing suppressorの例
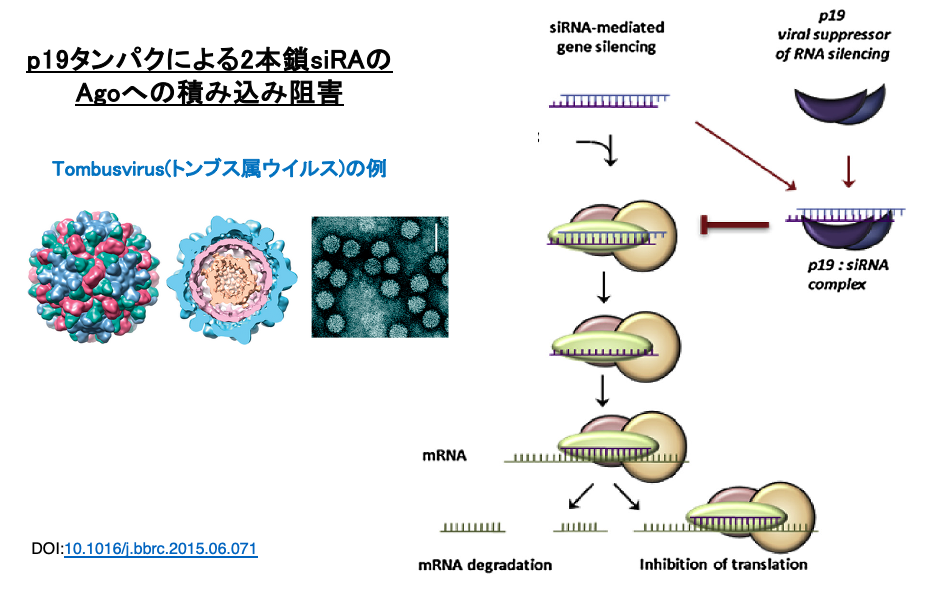
植物ウイルスであるTombusvirus属のウイルスではp19タンパクの2量体がsiRNA duplexに付着することで,Argonauteへのloadingを阻害します。これにより,植物側のRNA interferenceによるウイルスの増殖阻害作用を妨害しています。
29. 様々なウイルスRNAサイレンシングサプレッサーの作用点

30. Exosomeを介したmiRNAの伝搬
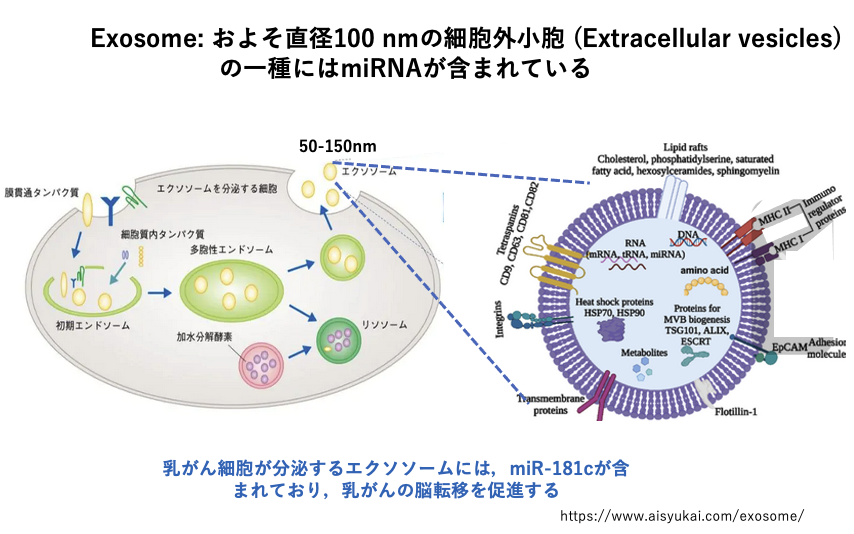
動物ではmiRNAが,exosomeとよばれる脂質2重膜を持つ小胞を介して,遠く離れた細胞に受け渡されることがあります。
Exosomeは,約100 nm径の分泌性の小胞で,血液やリンパ液で全身に運ばれることで,細胞間の長距離情報伝達が行われています。このexosomeには様々なタンパクや核酸が含まれていますが,miRNAも含まれています。
例えば乳ガン細胞が分泌するexosomeにはmicro RNA の一種であるmiR-181cが含まれており,これは乳ガンが脳に転移するのを促進する作用があることが分かっています。
31. piRNAによる転写後発現抑制の機構

次にpiRNA (PIWI-interacting RNA) による転写後発現抑制の機構について解説します。このpiRNA によるRNA interferenceは動物や昆虫の生殖細胞でのみ起こり,生殖細胞においるトランスポゾンの転移抑制に寄与しています。生殖細胞でのゲノム変異は,個体全体に影響を与え,進化の原動力にもなります。植物ではpiRNAは見つかっていません。
32. piRNA とmiRNAの比較
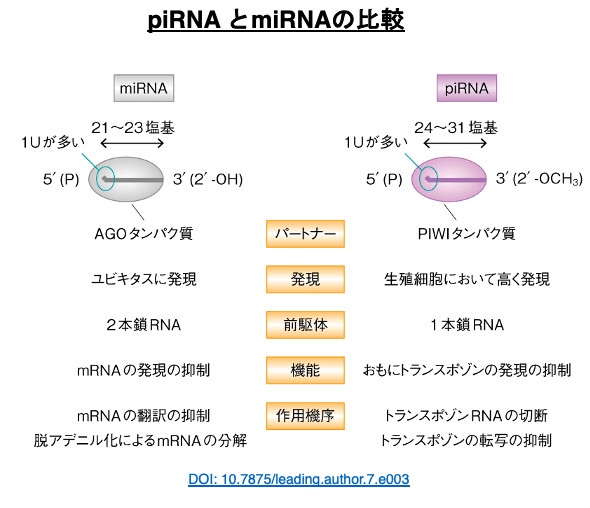
まずpiRNA (PIWI-interacting RNA)とmiRNAを比較してみます。その塩基長については,piRNAが24-31塩基とmiRNAの21-23塩基よりも長い傾向があります。また,組み込まれるタンパクはArgonauteではなく同系列のタンパクである PIWI (P-element induced wimpy testis)タンパクです。
このタンパクはArgonauteと同様にslicer活性を持っている場合があります。またpiRNAは生殖細胞において高く発現されます。miRNAの前駆体は2本鎖領域を持つヘアピンRNAですが,piRNAの前駆体は長い1本鎖RNAです。miRNAの標的mRNAは様々ですが,piRNAの標的はトランスポゾン由来のRNAです。miRNAは標的mRNAに対して結合して,分解を引き起こしますが,piRNAはトランスポゾン由来のRNAを切断します。
33. uni-strand piRNAクラスター
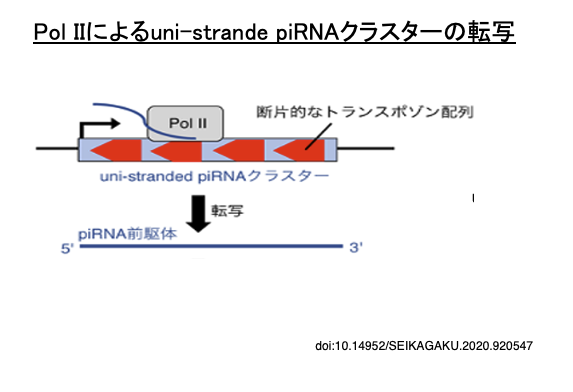
ショウジョウバエのゲノムには,様々なトランスポゾンの断片がセンス鎖・アンチセンス鎖という方向性に関して,同じ方向性で集まっている領域が知られています (uni-strand piRNA cluster)。
34. 活性化Piwi-piRISC の生成経路
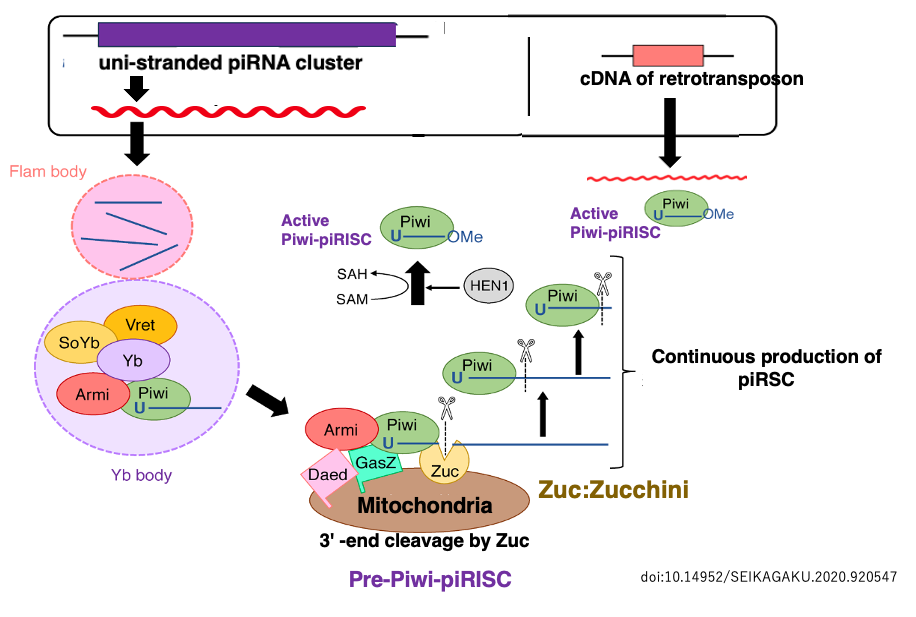
この長い1本鎖RNAは,Flam body に蓄積し,さらにYb bodyに移行して,そこで粗く切断されます。このPre-Piwi RNAはArgonauteと同系列のタンパクである PIWIに結合して,Pre-Piwi-piRISCを形成します。
ミトコンドリア膜上にあるエンドリボヌクレアーゼ Zucchini (野菜のズッキーニ,Zuc) がPiwi内にあるPre-piRNAの末端を切断することで24-31塩基長の成熟したpiRNAがPiwi内に形成されることで,成熟したPiwi-piRISCが生まれます。
残りの長いflamenco RNA(piRNA前駆体)は別のPIWIに捕獲され,先ほどと同様にZucによりその3’-末端が切断されるという,Dicer非依存的な反応が繰り返されます。その結果,1本鎖のpiRNA前駆体から大量の成熟したpiRNAが作られ,活性型Piwi-piRISCが生産されます。
活性化したPiwi-piRISCは,細胞質内でpiRNAと相補性をもつトランスポゾン由来のRNAを探索して発見すればそれを切断する,あるいは翻訳阻害を誘導します。
35. Dual-strand clusterの転写物から成熟piRNAが生産されるping pong経路
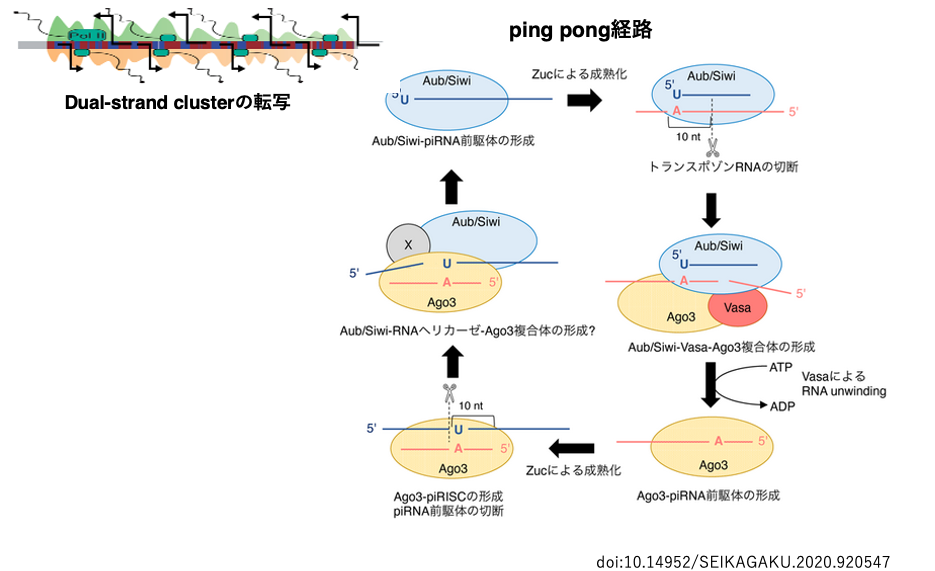
ショウジョウバエのflamenco領域にコードされているトランスポゾンの断片には方向性がり,その転写物はトランスポゾンのアンチセンス配列となるので,piRNA前駆体として適していました。
一方,ショウジョウバエのセントロメアやトロメア近傍のヘテロマチン領域(42AB, 38C, 80Fなど)には,トランスポゾン断片がセンス・アンチセンスという方向性がバラバラにコードされている領域があります。この領域については,ゲノムの両方からの転写が起こるのでdual-strand clusterと呼ばれます。この領域の両鎖の転写物は,一方の鎖から作られたpiRNAが他方の鎖の転写物からpiRNAを生産するために使用されます。そのため,このようなpiRNAの生産経路はping pong(ピンポン)経路と呼ばれます。
piRNAがPIWIタンパクに内蔵されてactiveなPiwi-piRISCとなるのは,uni-strand piRNA clusterからpiRNAが生産された場合と同じです。このようにsiRNAやmiRNAの生産にはDicerが関与しますがpiRNAの生産には,Dicerが関与しません。
では,これで終わります。
コメント